Author: haoransun
Wechat: SHR—97
论文:📎s41746-022-00577-x.pdf
补充材料:📎41746_2022_577_MOESM1_ESM.pdf
报告摘要:📎41746_2022_577_MOESM2_ESM.pdf
文章官方网址:https://www.nature.com/articles/s41746-022-00577-x#MOESM1
录用:NPJ | digital medicine SCI医学1区
1-摘要
现状:ADNI领域数据集有限。
问题:小数据集上的模型会过拟合,且未进行跨组评估。
解决思路:为了抑制有限数据导致的过拟合,作者提出一种混合机器学习框架,由多个CNN组成,引入大脑先验知识,从脑段角度自动提取图像特征(根据临床发现,这些特征确实与认知衰退有关)。
最终分类器:采用Linear-SVM,将上面提取到的图像特征与非图像信息进行结合做最终的预测。
模型性能:与其他最先进的方法相比,模型具有优越的性能,且模型具有高度的概括性(因为进行了跨组评估),可泛化于不用人种之间。
2-模型架构
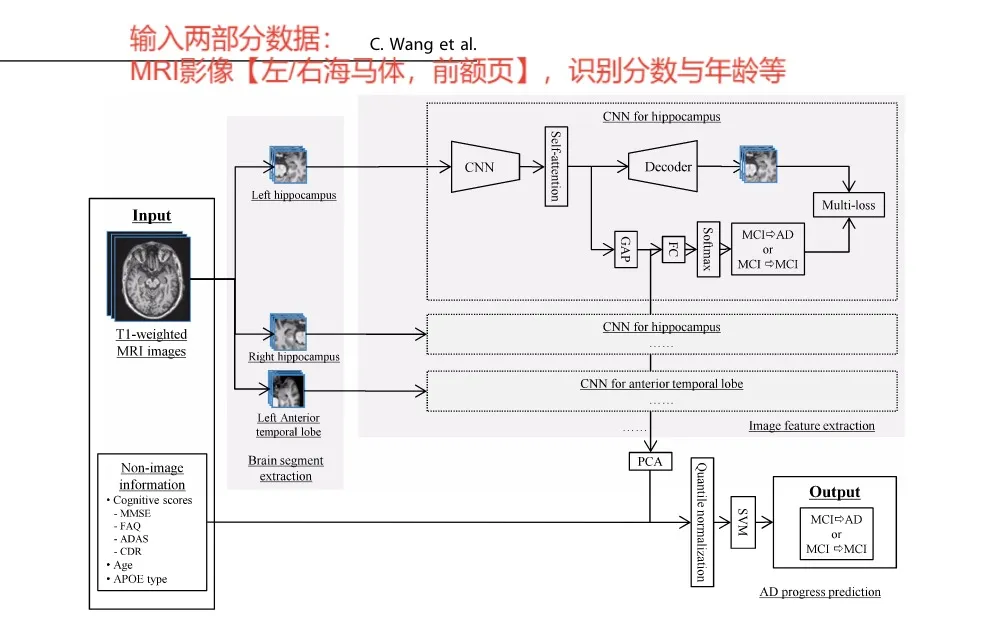
3-数据集
尽管PET和脑脊液(CSF)检查对AD诊断更有用,但由于其成本高,未被选中.
使用的是相对容易获得的信息作为输入,如MRI和非图像信息。
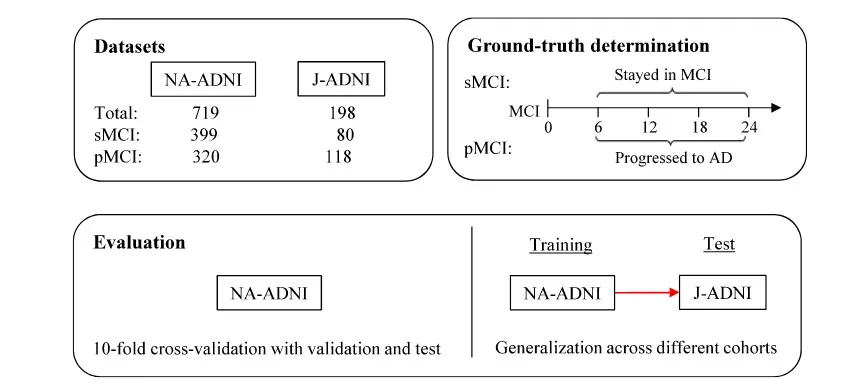
数据来源大部分都是北美白人队列,该数据集的样本数量相对较少,因此有必要使用完全不同的数据集评估训练后的模型(缺乏不同人种之间关于AD的横向对比),为了使该模型泛化性能更强,作者采用了两套数据来做实验。因为有研究表明:高加索人与东亚人之间的大脑皮层结构存在差异**。**
NA-ADNI:北美白人ADNI人群作为训练/验证模型
J-ADNI: 东亚(日本)作为测试模型
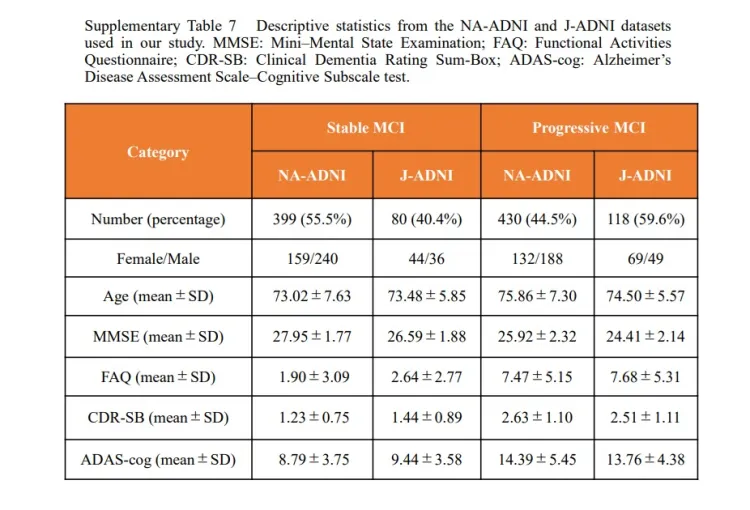
补充表7:NA-ADNI和J-ADNI具体数据
4-工具
SmoothGrad可视化:研究IFEP中CNN关注的领域。
A:整个大脑作为输入的可视化。
B:脑段作为输入的可视化。
B比A更关注于海马体和杏仁核。
红色表明对AD研究贡献大,蓝色表明对AD研究贡献小。

UMAP:研究模型提取特征的有效性,将训练样本的图像特征和非图像特征投影到二维(2D)空间,图5如下。
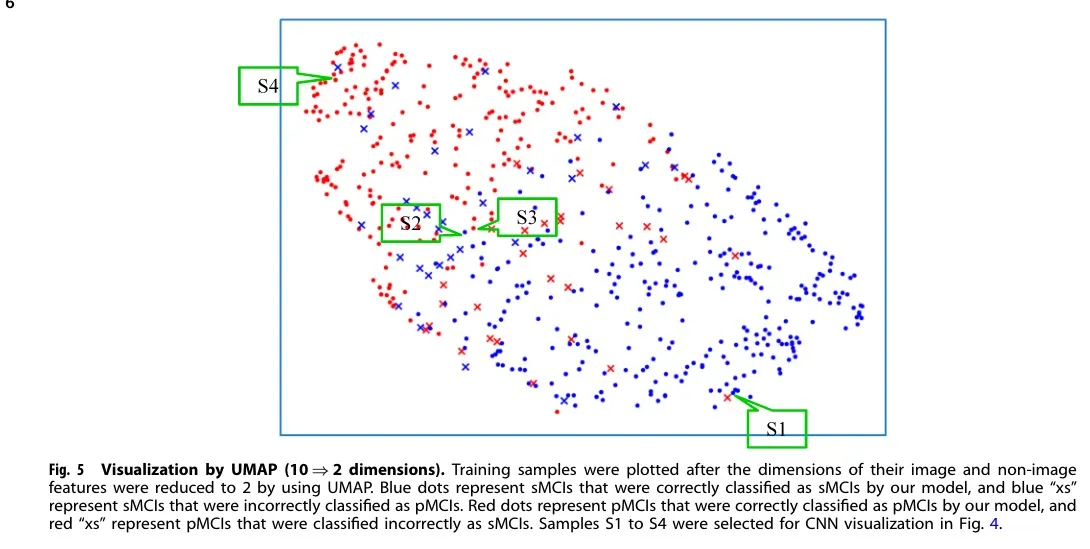
使用UMAP将训练样本的图像和非图像特征的尺寸减少到2后,绘制训练样本。
蓝色圆点表示我们的模型正确归类为sMCI的sMCI,
蓝色“xs”表示错误归类为pMCI的SMCI。
红点圆点表示我们的模型正确分类为pMCI的pMCI,
红色“xs”表示错误分类为sMCI的PMCI。
结论:****尽管存在少量的误分类错误,但大多数训练样本都是适当分布的,这表明成功地提取了用于分类sMCI和pMCI的有效特征,并且线性SVM适合于分类任务。
5-挑战
挑战1:如何通过使用有限数量的训练样本,有效的从图像中提取有意义的特征?
使用CNN,但有可能会因为样本过少而过拟合陷入局部最优解中,导致对未知数据的泛化性能变差。
引入脑成像先验知识,在IFEP中,不针对整个大脑做CNN,而是针对从全脑图像中裁剪的不同脑段训练多个CNN,最终选择了与AD高度相关的海马体和颞叶前部。通过采用脑段,每个样本的输入图像尺寸减少到不到整个大脑的十分之一,另外,每个CNN只能与AD进展高度相关的区域学习图像特征。
挑战2:采取哪种分类器作为最终预测?
既要兼顾IFEP中提取的图像特征,又要兼顾非图像信息,此论文选用了泛化性能更高的SVM。
6-消融实验
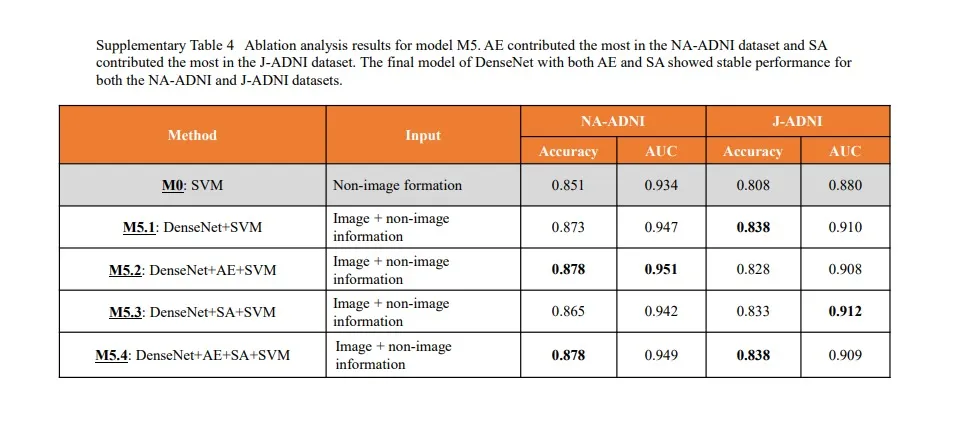
主干架构采用DenseNet,消融AE(Auto-Encoder)和SE(Self-Attention),论文表明,AE在NA-ADNI数据集中贡献大,SA在J-ADNI数据集中贡献大。两者都存在的DenseNet模型在这两个数据集上性能稳定。
补充表4
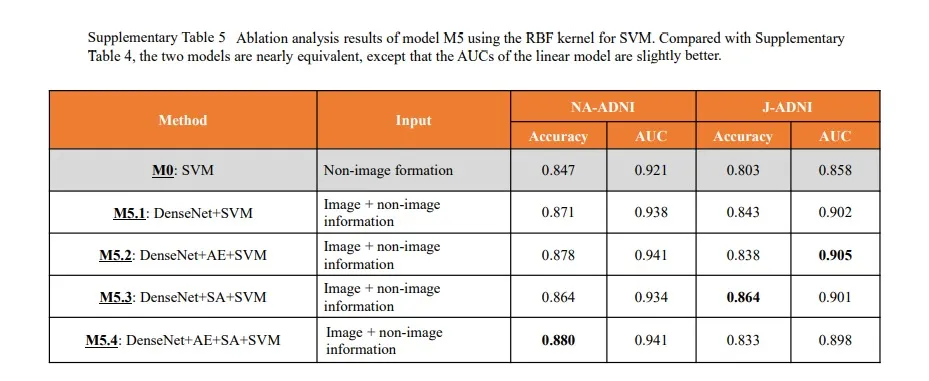
补充表5:使用RBF SVM进行消融实验对比
SVM中的SVC()算法包含两种核函数
- SVC(kernel = ‘ploy’):表示算法使用多项式核函数,依靠升维使得原本线性不可分的数据线性可分。
- SVC(kernel = ‘rbf’):表示算法使用高斯核函数,依靠降维度使得非线性可分的样本可分。
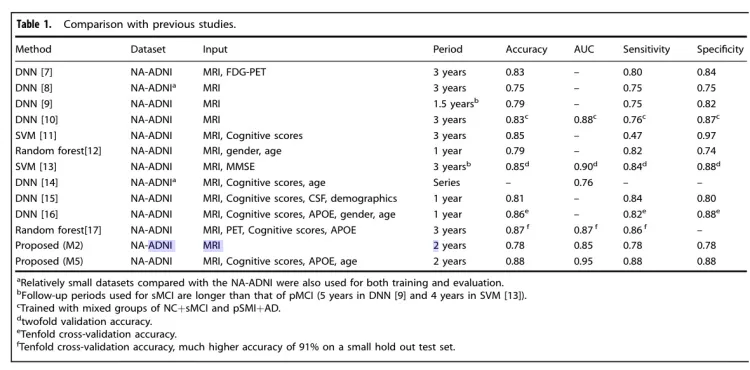
7-对比实验
表1:作者实验结果和以往其他人实验结果对比(突出作者模型优秀)
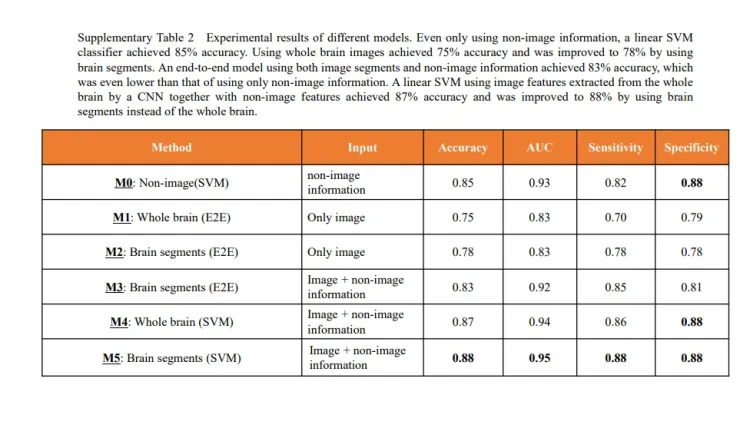
补充表2:SVM和E2E在不同输入信号上的实验结果对比(证明为什么选用SVM而不选用E2E)
E2E:端到端指的是输入的是原始数据,输出是最后的结果。
原来输入端不是直接的原始数据,而是在原始数据中提取的特征,这一点在图像问题上尤为突出,因为图像像素数太多,数据维度高,会产生维度灾难,所以原来一个思路是手工提取图像的一些关键特征,这实际就是就一个降维的过程。
那么问题来了,特征怎么提?
特征提取的好坏异常关键,甚至比学习算法还重要,举个例子,对一系列人的数据分类,分类结果是性别,如果你提取的特征是头发的颜色,无论分类算法如何,分类效果都不会好,如果你提取的特征是头发的长短,这个特征就会好很多,但是还是会有错误,如果你提取了一个超强特征,比如染色体的数据,那你的分类基本就不会错了。
这就意味着,特征需要足够的经验去设计,这在数据量越来越大的情况下也越来越困难。 > 于是就出现了端到端网络,特征可以自己去学习,所以特征提取这一步也就融入到算法当中,不需要人来干预了
经典机器学习方式是以人类的先验知识将raw数据预处理成feature,然后对feature进行分类。分类结果十分取决于feature的好坏。所以过去的机器学习专家将大部分时间花费在设计feature上。那时的机器学习有个更合适的名字叫feature engineering 。
后来人们发现,利用神经网络,让网络自己学习如何抓取feature效果更佳。于是兴起了representation learning。这种方式对数据的拟合更加灵活。
网络进一步加深,多层次概念的representation learning将识别率达到了另一个新高度。于是你听到了是个搞机器学习的人都知道的名字:deep learning。实指多层次的特征提取器与识别器统一训练和预测的网络。
end to end的好处:通过缩减人工预处理和后续处理,尽可能使模型从原始输入到最终输出,给模型更多可以根据数据自动调节的空间,增加模型的整体契合度。
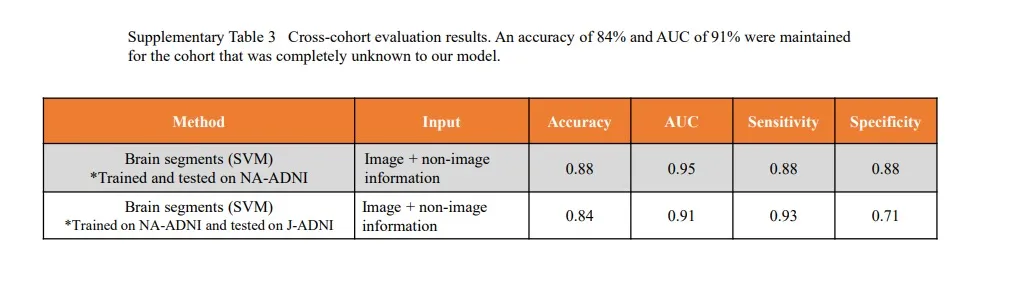
补充表3:模型在NA-ADNI和J-ADNI人种之间的实验结果对比(突出作者模型泛化能力强,可适配不同人种)
补充表6:最先进的方法仅通过验证数据集(而不是测试数据集)进行评估(作者模型的潜在优势应该更大)
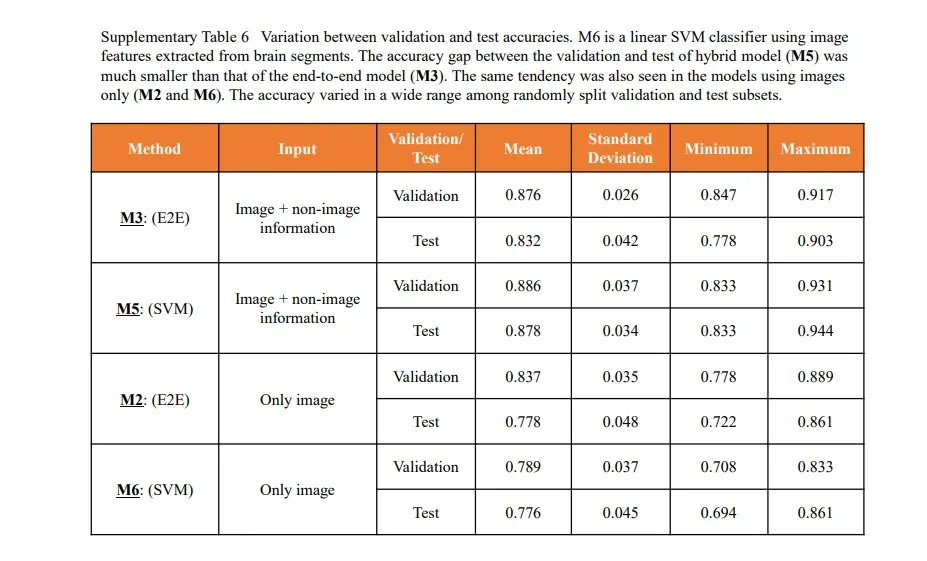
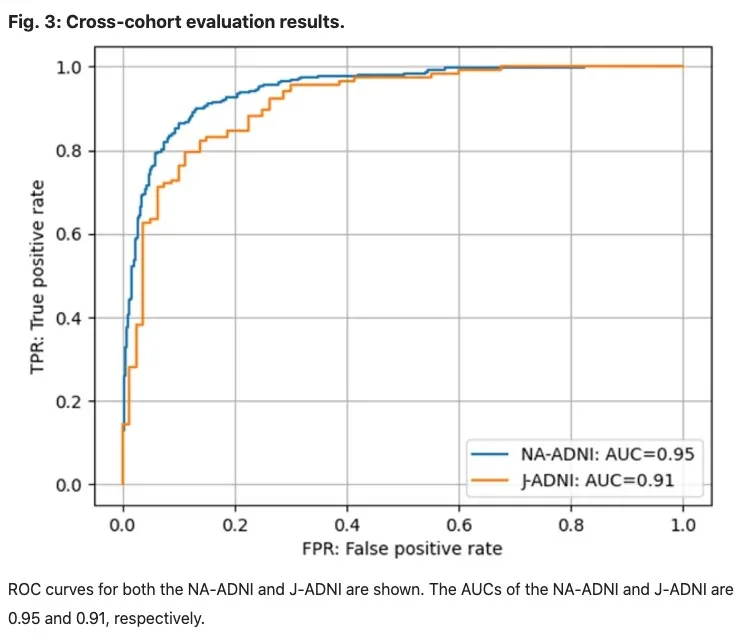
图3:不同人种之间的评估结果:AUC越高,模型检测真实率越高。
8-局限性
局限1:数据集小
尽管ADNI数据集是世界上关于AD最大的数据库,但其规模仍然相对较小,作者提出混合模型来抑制小数据上的过拟合。
局限2:数据利用率不高
作者的目标是为临床试验做贡献,仅采用基线数据作为输入,为了将其推广到可以获得纵向数据的常见临床诊断,不仅使用基线数据,还要利用好整个纵向数据,有待改进。
局限3:数据在动态发展,现有模型只是当前表现优越
由于数据集过小,作者提出的混合模型比使用主流的DenseNet作为主干的端到端模型显得比较优越,但ADNI数据集在逐年增大中,最先进的架构(如视觉转换器)作为主干模型应当继续研究。
9-ADNI简介
官方网址:https://adni.loni.usc.edu/
作者用于训练、验证、测试的数据集来自于ADNI1/ADNI-GO/ADNI2。
NA-ADNI是一项队列研究,以公私合作的形式由首席研究员Michael W.Weiner M.D发起。
NA-ADNI研究招募了2000+认知正常的参与者和MCI或AD患者。
ADNI-1:200CN个体,400晚期MCI患者,200名轻度痴呆患者,总共800人。
ADNI-GO:约200+早期MCI患者。
ADNI-2:招募了更多处于AD不同阶段的参与者进行AD监测。
ADNI-3:由于正在收集更多患者数据中,目前没有可用的诊断信息,未被作者采纳。
10-参与者筛选
每个参与者都要进行纵向数据的筛查:首先确定参与者被诊断为MCI的候选基线点,并且T1加权MRI图像和该患者的其他非图像信息均可用。
非图像信息包括简易精神状态检查(MMSE)、功能活动问卷(FAQ)、临床痴呆症评分(CDR)盒子总和评分、阿尔茨海默病评估量表Cog-11(ADAS)、APOE类型(载脂蛋白E)和人口统计信息,如性别和年龄。
如果参与者在基线检查后的20年内发展为AD,我们将该参与者与基线检查时的T1加权MRI图像和非图像信息一起标记为pMCI样本;否则,作为sMCI样本。
实验提取了399个sMCI和430个pMCI样本,用于训练、验证和测试作者的预测模型。同样的程序应用于J-ADNI数据集,从J-ADNI数据集中提取了总计80个稳定的和118个进行性MCI样本,用于评估我们的训练模型。
11-图像处理
图像形状规范化
研究中使用的3D T1加权MRI图像首先通过将T1加权MRI图像与在MNI空间创建的图谱图像(标准图像)对齐,转换为蒙特利尔神经研究所(MNI)空间。使用了MNI152 NLIN 2009a地图集的模板图像。
图像强度归一化
由于使用不同设备在不同地点获取的T1加权MRI图像在强度分布方面可能有不同的偏差,因此图像强度归一是许多图像分析任务的重要预处理。为了确保用于强度归一化的形状归一化andatas模板图像中对应的斑块包含相同的组织,我们在形状归一化andatas模型图像和atlas模板图像之间进行了非线性图像配准。
最后基于ITK实现了非线性图像配准。
脑段提取
在进行强度归一化后,对形状归一化图像执行颅骨剥离过程,以提取大脑区域。训练了一个四层V-net。V-net使用与地标检测相同的数据集进行训练。训练数据的大脑区域的是在放射技师的监督下通过手工编辑获得的。分割结果由图像配准方法自动生成,该方法还包括在ITK中实现的不同地貌图像配准。从标准化(形状和强度)和颅骨断层图像中,提取海马(左和右)和前颞叶(左和右侧)的脑段。这些片段的位置是在影像模板图像中手动确定的,它们的大小固定为64×64×64像素,足够大,可以包含标准化图像中每个感兴趣的片段,并有必要的边界。对于每个样本,分别从其归一化图像中提取出位于图谱模板图像中指定位置的四个脑段,其中包含海马和颞前叶,左右两侧。
图像特征提取
DenseNet作为提取图像特征的主干网络,引入SA和AE来增强提取效果。
12-Linear SVM Classification
由于训练样本有限,作者采用线性SVM替代E2E deep学习作为最终预测,提高泛化能力。
通过CNN从每个脑段提取128维图像特征后,使用PCA将每个脑段维数降为1。然后将非图像信息,如认知得分(MMSE/FAQ/CDR/ADAS)年龄、APOE类型,与四个片段的简化图像特征结合,作为 SVM的输入。
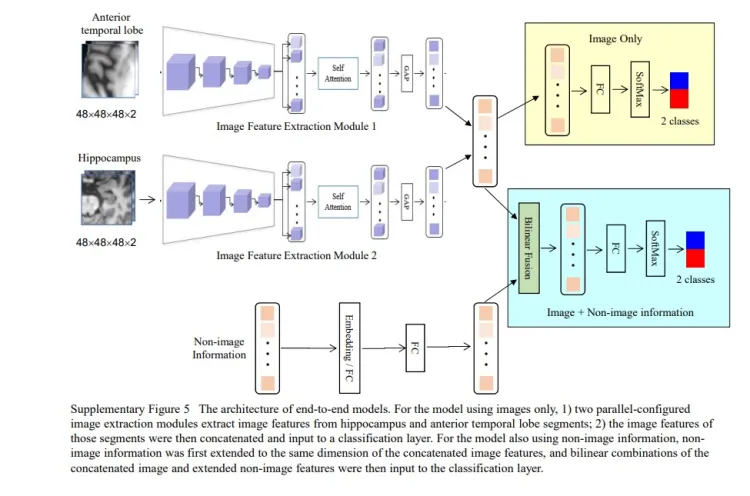
13-End-to-End(E2E) Classification
作为对比实验采用实现了两个E2E模型,一个仅使用图像进行预测,另一个同时使用图像和非图像信息。
对于同时使用图像和非图像信息,双线性融合(Bilinear fusion)用于组合从图像中提取的图像特征和从非图像中提取的非图像特征。作者配置了两个图像特征提取模块,并行的从海马和前颞图像中提取图像特征,并将图像特征连接在一起。
作者首先嵌入非图像特征,然后被扩展到与图像特征相同的维度。然后将双线性融合应用得到图像特征和扩展后的非图像特征,从而生成用于分类的多模态特征。

14-DATA AVAILABILITY
NA-ADNI dataset:https://adni.loni.usc.edu/
J-ADNI dataset:https://humandbs.biosciencedbc.jp/en/hum0043-v1)
15-CODE AVAILABILITY
The DenseNet code, which was used as the backbone of our architecture, is available at
https://github.com/liuzhuang13/DenseNet
主干网络:提取图片特征
The Faster R-CNN code used for brain landmark detection is available at
https://github.com/rbgirshick/py-faster-rcnn
目标检测模型:脑段抽取
The Insight Toolkit: ITK, on which our image registration algorithm was implemented, is available at
医学影像处理工具:图像配准
16-文献参考及引用缘由
作者为什么研究这个课题->目前这个这个课题的研究现状->作者想解决什么问题->作者通过什么理论/模型解决这个问题->作者给出的实验结果如何->作者关于当前模型的不足与未来猜想
- Alzheimer’s Association. Alzheimer’s disease facts and figure. Alzheimer’s Dement. 15, 321–387 (2019). 2019.
(在Introduction中,开篇阐述了AD的症状和给社会带来的负担,调研了北美AD患者在2019年的数量,预测了2050年的AD患者数量,AD患者数量剧增,研究AD发展很有意义)
- Cummings, J., Lee, G., Ritter, A., Sabbagh, M. & Zhong, K. Alzheimer’s disease drug development pipeline: 2019. Alzheimer’s Dement. 5, 272–293 (2019).
- Cummings, J., Lee, G., Ritter, A., Sabbagh, M. & Zhong, K. Alzheimer’s disease drug development pipeline: 2020. Alzheimer’s Dement. 23, 1–24 (2021).
(AD治疗方案有限且失败率高,AD药物研究发展缓慢,新药研发面临挑战,大部分药物研发的目的是减缓早期AD的病变速度)
- Manly, J. J. et al. Frequency and course of mild cognitive impairment in a multiethnic community. Ann. Neurol. 63, 494–506 (2008).
- Petersen, R. C. et al. Practice guideline update summary: mild cognitive impairment. Neurology 16, 126–135 (2018).
(MCI患者是临床诊断的主要对象,那些被招募的MCI患者中,重点关注会从MCI演变为AD的患者,因为有20%的MCI在1.5-2年后被确诊为AD,80%的MCI稳定或者返回CN)
- Mueller, S. G. et al. The Alzheimer’s disease neuroimaging initiative. Neuroimaging Clin. North Am. 15, 869–877 (2005).
(AD患者数据大部分来自于NA-ADNI)
- Lu, D., Popuri, K., Ding, G. W., Balachandar, R. & Beg, M. F. Multimodal and multiscale deep neural networks for the early diagnosis of Alzheimer’s disease using structural MR and FDG-PET images. Sci. Rep. 8, https://doi.org/10.1038/s41598-018-22871-z (2018).
- Basaia, S. et al. Automated classification of Alzheimer’s disease and mild cognitive impairment using a single MRI and deep neural networks. NeuroImage: Clin. 21, https://doi.org/10.1016/j.nicl.2018.101645 (2019).
- Zhang, J. et al. A 3D densely connected convolution neural network with connection-wise attention mechanism for Alzheimer’s disease classification. Magn. Reson. Imaging 78, 119–126 (2021).
- Abrol, A. et al. Deep residual learning for neuroimaging: An application to predict progression to Alzheimer’s disease. J. Neurosci. Methods 339, https://doi.org/10.1016/j.jneumeth.2020.108701 (2020).
(7-10模型仅使用脑图信息作为输入)
- Tam, A. et al. A highly predictive signature of cognition and brain atrophy for progression to Alzheimer’s dementia. GigaScience 8, https://doi.org/10.1093/gigascience/giz055 (2019).
- Ledig, C., Schuh, A., Guerrero, R., Heckemann, R. A. & Rueckert, D. Structural brain imaging in Alzheimer’s disease and mild cognitive impairment: biomarker analysis and shared morphometry database. Sci. Rep. 8, https://doi.org/10.1038/s41598-018-29295-9 (2018).
- Syaifullah A. et al. Machine learning for diagnosis of AD and prediction of MCI progression from brain MRI using brain anatomical analysis using diffeomorphic deformation. Front. Neurosci. 11, https://doi.org/10.3389/fneur.2020.576029 (2021).
- Nakagawa, T. et al. Prediction of conversion to Alzheimer’s disease using deep survival analysis of MRI images. Brain Commun. 2, https://doi.org/10.1093/braincomms/fcaa057 (2020).
- Lee, G., Nho, K., Kang, B., Sohn, K. A. & Kim, D. Predicting Alzheimer’s disease progression using multi-modal deep learning approach. Sci. Rep. 9, https://doi.org/10.1038/s41598-018-37769-z (2019).
- Goto, T., Wang, C., Li, Y. & Tsuboshita, Y. Multi-modal deep learning for predicting progression of Alzheimer’s disease using bi-linear shake fusion. Proc. SPIE (Med. Imaging) 11314, 452–457 (2020).
- El-Sappagh, S. et al. A multilayer multimodal detection and prediction model based on explainable artificial intelligence for Alzheimer’s disease. Sci. Rep. 11, https://doi.org/10.1038/s41598-021-82098-3 (2021).
(11-17模型使用多模态信息:图像和认知分数等非图信息)
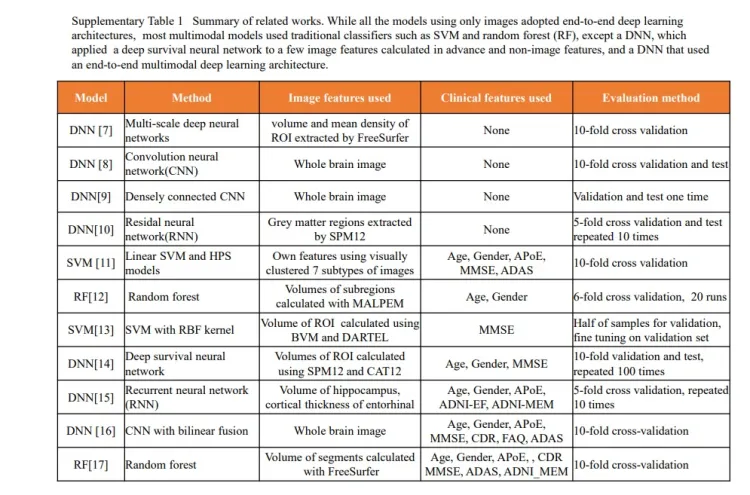
一些已提出的相关工作在补充表1中提出,如下图(补充表1)
- Srivastava, N., Hinton, G., Krizhevsky, A., Sutskever, I. & Salakhutdinov, R. Dropout: a simple way to prevent neural networks from overfitting. J. Mach. Learn. Res. 15, 1929–1958 (2014).
(大部分已知模型使用的是深度学习,这需要大量的训练样本和可调节的参数,引入Dropout来让模型达到一个好的泛化性能)
- Wang, X. et al. ChestX-Ray8: Hospital-Scale Chest X-Ray Database and benchmarks on weakly-supervised classification and localization of common thorax diseases. Proc. IEEE Conf. on Computer Vision and Pattern Recognition (CVPR), 3462-3471 (2017).
- Deng, J. et al. ImageNet: A large-scale hierarchical image database. Proc. IEEE Conf. on Computer Vision and Pattern Recognition (CVPR), 248-255 (2009).
(尽管sMCI和pMCI分类准确率达到了75%-86%之间,但与上述两个数据集相比,仍然存在问题。第一个问题是ADNI数据量过小,只有成千上百个,容易导致过拟合)
- Iwatsubo, T. et al. Japanese and North American Alzheimer’s disease neuroimaging initiative studies: harmonization for international trials. Alzheimer’s Dement. 14, 1077–1086 (2018).
- Kang, D. W. et al. Differences in cortical structure between cognitively normal East Asian and Caucasian older adults: a surface-based morphometry study. Sci. Rep. 10, https://doi.org/10.1038/s41598-020-77848-8 (2020).
(第二个问题是由于患者数据的偏见,即AI模型仅在高加索人人种范围内进行训练和评估,模型的实际能力受到了制约,训练的AI模型有必要在其他人种队列中进行测试,以验证模型的泛化性能。据研究表明,北美白人和东亚人在大脑皮层结构中有差异,这类不同人种队列的方法还没有被使用,作者在这篇文章中提出了此方法 ,创新点)
- LeCun, Y., Bengio, Y. & Hinton, G. Deep learning. Nature 521, 436–444 (2015).
- Yamaguchi, H. et al. Three-dimensional convolutional autoencoder extracts features of structural brain images with a “diagnostic label-free” approach: application to Schizophrenia Datasets. Front. Neurosci. 07, https://doi.org/10.3389/fnins.2021.652987 (2021).
(在IFEP中,采用经典的CNN架构自动提取图像特征)
- Wang, L. et al. Alterations in cortical thickness and white matter integrity in mild cognitive impairment measured by whole-brain cortical thickness mapping and diffusion tensor imaging. Am. J. Neuroradiol. 30, 893–899 (2009).
- Misra, C., Fan, Y. & Davatzikos, C. Baseline and longitudinal patterns of brain atrophy in MCI patients, and their use in prediction of short-term conversion to AD: Results from ADNI. Neuroimage 44, 1415–1422 (2009).
- Risacher, S. L. et al. Baseline MRI predictors of conversion from MCI to probable AD in the ADNI cohort. Curr. Alzheimer Res. 6, 347–361 (2009).
(在IFEP中,为了抑制深度学习所带来的过拟合,作者引入脑成像先验知识,请大脑领域研究员挑选与认知下降高度相关的脑段来进行研究,减少图像维度)
- Shorten, C. & Khoshgoftaar, T. A survey on image data augmentation for deep learning, J. Big Data, 6, 1–48, (2019).
- Vaswani, A. et al. Attention is all you need. Proc. Int’l Conf. on Neural Information Processing Systems, 6000–6010 (2017).
- Vincent, P., Larochelle, H., Bengio, Y. & Manzagol, P. A. Stacked denoising autoencoders: learning useful representations in a deep network with a local denoising criterion. J. Mach. Learn. Res. 11, 3371–3408 (2010).
- Hinton, G. E., Krizhevsky, A. & Wang, S. D. Transforming auto-encoders. Proc. Int’l Conf. on Artificial Neural Netw. 44-51 (2011).
(为了提高CNN的性能和泛化能力,作者引入了数据增强、18-Dropout、SA、AE)
- Brereton, R. G. & Lloyd, G. R. Support Vector Machines for classification and regression. The Analyst Online 135, https://doi.org/10.1039/B918972F (2010).
- Girshick, R., Donahue J., Darrell, T. & Malik, J. Rich feature hierarchies for accurate object detection and semantic segmentation. Proc. IEEE Conf. on Computer Vision and Pattern Recognition (CVPR), 580-587 (2014).
(线性SVM比非线性SVM有更高的泛化能力,使用它作为分类器对CNN提取的图像特征和非图像特征进行分类效果更佳)
- Iwatsubo, T. Japanese Alzheimer’s disease neuroimaging initiative: present status and future. Alzheimer’s Dement. 6, 297–299 (2010).
(J-ADNI数据集)
- Smilkov, D., Thorat, N., Kim, B., Viégas, F. & Wattenberg, M. SmoothGrad: removing noise by adding noise, Proc. Int’l Conf. on Machine Learning (ICML), https://doi.org/10.48550/arXiv.1706.03825 (2017).
(SmoothGrad:有待进一步了解)
M2和M5表明脑段作为输入优于全脑输入M1和M4。

(为了可视化在IFEP部分CNN关注的区域,使用了SmoothGrad工具进行可视化)
上述脑段输入优于全脑输入的原因有下图给出,ROI区域被重点关注,模型精力不会被分散.

S1-S4样本的选择来自于补充图5(颞叶前部和海马体由有先验知识的脑专家手动指定)
- McInnes, L., Healy, J., Saul, N. & Großberger, L. UMAP: Uniform Manifold Approximation and Projection. J. Open Source Softw. 3, 861. https://doi.org/10.21105/joss.00861 (2018).
(UMAP工具,有待进一步了解)
- Rajkomar, A., Dean, J. & Kohane, I. Machine learning in medicine. N. Engl. J. Med. 380, 1347–1358 (2019).
(深度学习模型的优点有时也会是其脆弱性之一,既数据量小时容易陷入局部最优解)
- Buolamwini, J. & Gebru, T. Gender shades: intersectional accuracy disparities in commercial gender classification. Proc. Mach. Learn. Res. 81, 1–15 (2018).
(性别识别系统中报告了由队列(北美和东亚)和性别偏见造成的意外影响)
- Dosovitskiy, A. An image is worth 16x16 words: transformers for image recognition at scale. Proc. Int’l Conf. on Learning Representations (ICLR), https://arxiv.org/abs/2010.11929 (2021).
(由于ADNI数据量有限,作者的模型表现优越,但随着ADNI的不断发展,先进的模型也会变的更加优越,比如这里引用的视觉转换模型)
- Palmqvist, S. et al. Prediction of future Alzheimer’s disease dementia using plasma phospho-tau combined with other accessible measures. Nat. Med. 27, 1034–1042 (2021).
(新的研究表明,血浆磷-τ,对预测也有效,有必要考虑在未来的模型中加入这些生物标记物)
- Brett, M., Johnsrude, I. S. & Owen, A. M. The problem of functional localization in the human brain. Nat. Rev. Neurosci. 3, 243–249 (2002).
- Fonov, V. S., Evans, A. C., McKinstry, R. C., Almli, C. R. & Collins, D. L. Unbiased nonlinear average age-appropriate brain templates from birth to adulthood. NeuroImage 47, 539–541 (2009).
(将T1加权MRI图像与在MNI空间创建的图谱图像(标准图像)对齐,首次将我们研究中使用的3D T1加权核磁共振图像转换为蒙特利尔神经研究所(MNI)空间。使用了MNI152 NLIN 2009a atlas的模板图像。为了将根据NA-ADNI或J-ADNI研究中T1加权MRI图像与在MNI空间中创建的atlas模板图像进行可靠、准确的对齐,开发了一种包含基于地标和基于图像配准的对齐步骤的粗到细方法)
- Ren, S., He, K., Girshick, R. & Sun, J. Faster R-CNN: Towards Real-Time Object Detection with Region Proposal Networks. Proc. IEEE Conf. on Computer Vision and Pattern Recognition (CVPR), 1137-1149 (2016).
- Marcus, D. S. et al. Open access series of imaging studies (OASIS): cross-sectional MRI data in young, middle aged, nondemented, and demented older adults. J. Cogn. Neurosci. 19, 1498–1507 (2007).
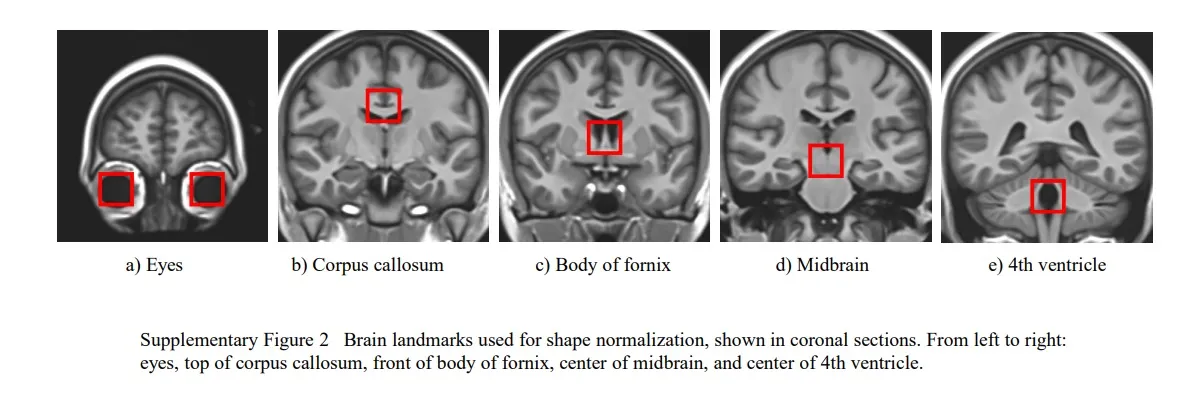
(基于区域的Fater R-CNN模型和OASIS数据集进行训练,训练图像的地标是在放射技师的监督下手动指定的,landmask为:左右眼,胼胝体(顶部)、穹窿体(前部)、中脑(中部)、第四脑室(中部),整体用于基于Landmask进行对齐)
- Maes, F., Vandermeulen, D. & Suetens, P. Medical image registration using mutual information. Proc. IEEE 91, 1699–1722 (2003).
- Oliveira, P. P. M. & Tavares, M. R. R. Medical image registration: a review. Computer Methods Biomech. Biomed. Eng. 17, 73–93 (2014).
- Mattes, D., Haynor, D. R., Vesselle, H., Lewellen, T. K. & Eubank, W. Nonrigid multimodality image registration. Proc. SPIE 4322, Medical Imaging: Image Processing, 1609-1620(2001).
(基于图像配准的对齐步骤中,通过使用图像配准技术,利用之前的方法进行图像相似度度量)
- Avants, B. B. et al. The Insight ToolKit image registration framework. Front. Neuroinformatics 8, 1–13 (2014).
(使用了与图谱一起提供的图谱模板图像的大脑掩码,以确保图像注册仅在大脑区域进行。使用的图像注册技术是基于开源Insight Tool Kit(ITK)图像注册框架实现,ITK有待进一步了解)
- Reinhold, J.C., Dewey, B.E., Carass, A. & Prince, J.L. Evaluating the impact of intensity normalization on MR image synthesis. Proc. SPIE 10949, Medical Imaging: Image Processing, https://doi.org/10.1117/12.2513089 (2019).
- Roy, S. & Carass, A. Magnetic resonance image example-based contrast synthesis. IEEE Trans. Med. Imaging 32, 2348–2363 (2013).
- Shinohara, R. T. et al. Statistical normalization techniques for magnetic resonance imaging. Neuroimage: Clin. 6, 9–19 (2014).
- Sun, X. et al. Histogram-based normalization technique on human brain magnetic resonance images from different acquisitions. BioMedical Eng. OnLine 14, https://doi.org/10.1186/s12938-015-0064-y (2015).
(过去提出的图像强度归一化方法)
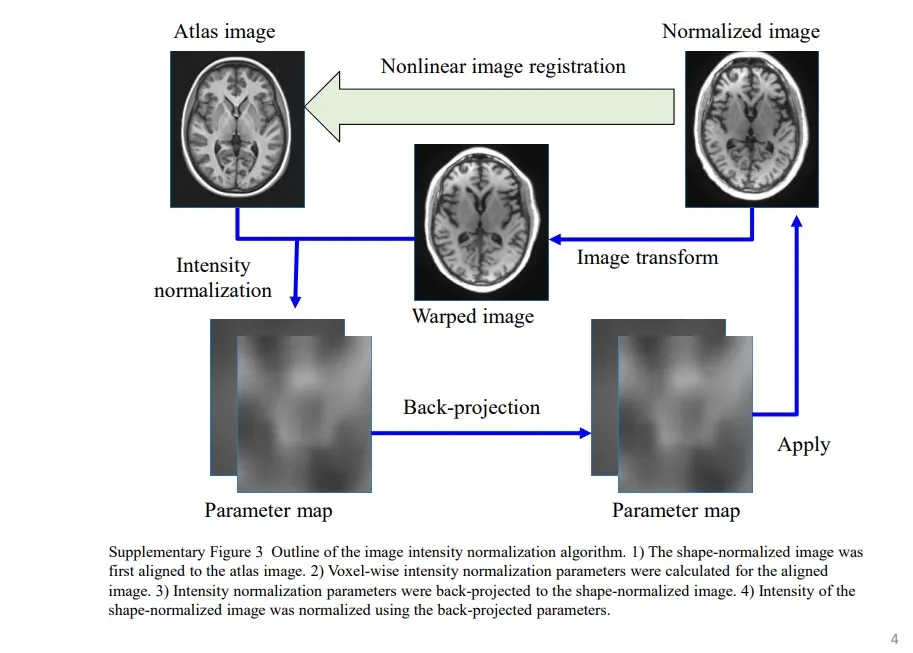
作者采用类似基于示例的强度归一化方法,该方法使用T1加权MRI图像和图谱模板图像中的斑块,其中包含所需的获得的斑块和组织对比度,但为了鲁棒性和计算简单性,作者以更简单的方式实现了它。
补充图3:显示了图像强度归一化算法的概要(有待进一步了解)
- Milletari, F., Navab, N. & Ahmadi, S. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Fourth Int’l Conf. on 3D Vision (3DV), 565-571 (2016).
(在进行强度归一化后,对形状归一化图像进行颅骨剥离处理,以提取大脑区域。作者训练了一个四层V-net)
- Ashburner, J. A fast diffeomorphic image registration algorithm. NeuroImage 38, 95–113 (2007).
(V-net使用与地标检测相同的数据集进行训练。训练数据的大脑区域的基本真相是在放射技师的监督下通过手工编辑获得的。分割结果由图像配准方法自动生成,该方法还包括在ITK中实现的差异图像配准)
- Huang, G., Liu, Z. & Maaten, L. Densely Connected Convolutional Networks. Proc. IEEE Conf. on Computer Vision and Pattern Recognition (CVPR), 4700-4708 (2017).
(作者采用的主干网络DenseNet模型)
- Islam, J. & Zhang, Y. Brain MRI analysis for Alzheimer’s disease diagnosis using an ensemble system of deep convolutional neural networks. Brain Informatics 5, https://doi.org/10.1186/s40708-018-0080-3 (2018).
(DenseNet在不同数据集上完成相同任务时比其他网络架构具有优势)
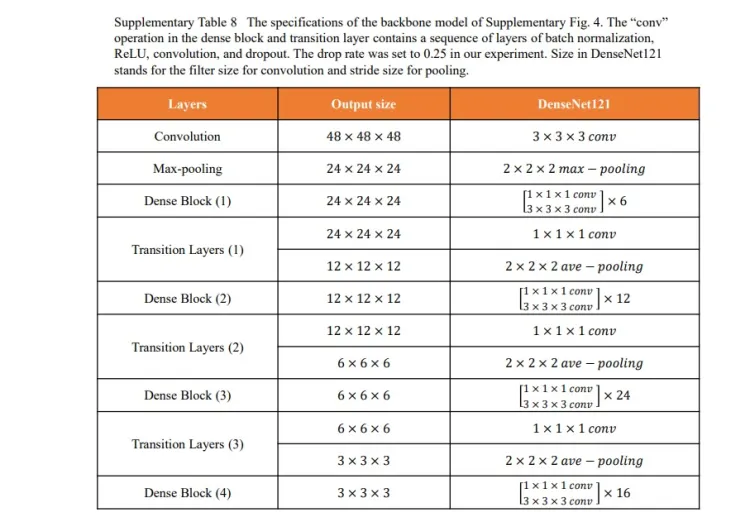
补充表8:显示了作者的DenseNet主干网的详细架构,该架构与DenseNet-121(参考文献55)基本相同,其中2D卷积层和池层被修改为3D层。由于脑段图像的尺寸较小,第一个卷积层的尺寸也变小了。
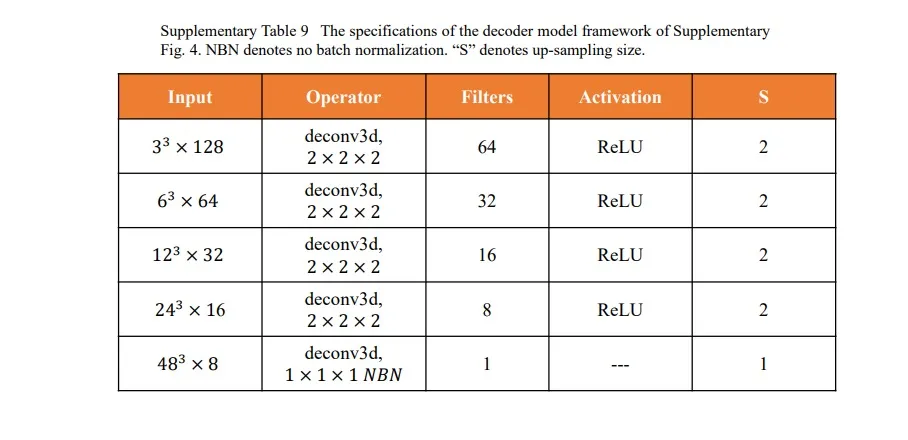
补充表9:除了将输入的脑段分类为sMCI或pMCI类的分类任务外,AE的另一个任务是从图像特征中恢复输入的脑片段图像,该任务被添加到DenseNet主干网中。通过引入AE任务,该任务被认为比使用少量样本的分类更健壮,预计整个体系结构的健壮性将得到提高。对于解码器,采用了一个简单的五层网络,由去卷积层和上采样层组成。
- Hsiunga, G-Y.R & Sadovnick, A.D. Genetics and dementia: risk factors, diagnosis, and management. Alzheimer’s Dement. 3, 418–427 (2007).
(根据该参考文献将APOE类型转换为代表AD风险的值)
- Bolstad, B. M., Irizarry, R. A., Astrand, M. & Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics 19, 185–193 (2003).
(由于这些多模态输入的分布差异很大,因此在应用SVM之前,使用四分位归一化组合特征进行归一化。为了进行验证和测试,将用于训练的相同四分位数标准化应用于组合特征)
18-有待了解与学习
- ITK:图像配准工具
https://github.com/InsightSoftwareConsortium/ITK
- UMAP:降为算法,可视化证明模型提取特征的有效性
https://doi.org/10.21105/joss.00861 (2018).
- SmoothGrad:证明脑段中与AD研究高度相关的区域(海马体、杏仁核、颞前叶)
https://doi.org/10.48550/arXiv.1706.03825 (2017).
- Faster R-CNN:基于区域的CNN,基于选取的六个Landmask进行图片对齐。
目标检测算法发展:R-CNN——Fast R-CNN——Faster R-CNN——Mask R-CNN
R-CNN:CVPR2014 https://arxiv.org/abs/1311.2524
Fast R-CNN:ICCV2015 https://arxiv.org/abs/1504.08083
Faster R-CNN:NIPS2015 https://arxiv.org/abs/1506.01497
Mask R-CNN:CVPR2017 https://arxiv.org/abs/1703.06870
- OASIS数据集:Open access series of imaging studies,训练Faster R-CNN
- DenseNet:主干网络,用于IFEP部分的图片特征自动提取https://openaccess.thecvf.com/content_cvpr_2017/html/Huang_Densely_Connected_Convolutional_CVPR_2017_paper.html
- APOE转为AD风险的算法
- 图像强度归一化算法:由于采集设备、采集环境的不同,需要对图像强度进行归一化
- 图像相似度度量算法:度量图像之间的相似度
- V-Net算法:基于3D卷积神经网络的方法,快速,准确的方式执行MRI体积的分割
https://github.com/mattmacy/vnet.pytorch
- 四分位归一化组合特征:特征工程归一化的方法之一