Author: haoransun
Wechat: SHR—97
1-横截面选取
Axial:又名横断面,transverseCoronal:又名冠状面,Sagittal:矢状面,如同一个箭矢劈开成左右两半
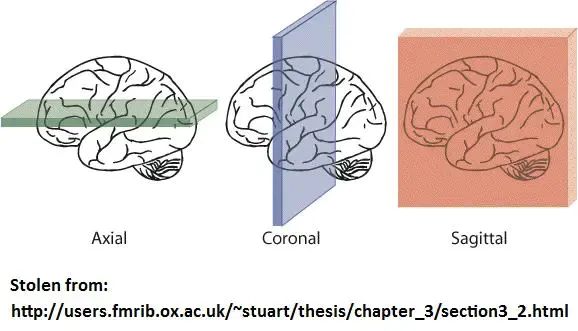
这种图就是横断面Axial
nii_process-origin.py
1 | import scipy, numpy, shutil, os, nibabel, glob |
2-同组内混合
1 | slices_AD_C = glob.glob("/home/pugongying/data/jupyterLab/shr/AD/ADNI-Datasets/AD/origin-slices/*/slice_C/*.png") |
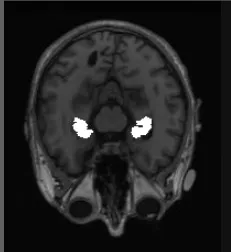
3-横截面 Axial进行方位校正,以便海马体的提取
1 | import pandas as pd |
4-UNet进行海马体分割
搭建UNet
1 | import torch |
模型对ADNI-Datasets 方向校正后的横截面数据作海马体预测提取
1 | import glob |
1 | class MRI_3_class_Dataset(Dataset): |
预训练好的模型在 阿里云 Unet中
1 | model = "/home/pugongying/data/jupyterLab/shr/AD/U-NET/model_version_2/model.pth" |
1 | AD_Data_Loader = load_mri_3_class_data(url="/home/pugongying/data/jupyterLab/shr/AD/ADNI-Datasets/AD/origin-slices_C_CD/*.png", batch_size=4, shuffle=True, split=None) |
5-海马体候选挑选
1 | 进入命令行, cp -r origin-slices_C_CD_hs/*.png origin-slices_C_CD_hs_ |
挑选颜色均值>2的L_R图片
1 | import os |
融合图片,将左右海马体融合到原图上
- 融合后原图放在origin-slices_C_CD_hs_Mix中
1 | def concat_iamge_hippocampus(url, target): |
6-补充AD-MCI
AD_Origin_Mix: 178
CN_Origin_Mix: 352
MCI_Origin_Mix: 215
所以需要再多下载AD和MCI 进行补充
补充后:
AD_Origin_Mix: 300
CN_Origin_Mix: 500
MCI_Origin_Mix: 380
FSL左右海马体分割+freesurfer合并
https://blog.csdn.net/qq_38851184/article/details/124452709
背景
需要分割出海马体。可借鉴方法:深度学习(UNet分割),形态学上的开闭,fsl中的分割。
使用fsl种的分割时需要注意,fsl分割分为左海马和右海马
方法:使用fsl中的first命令进行分割。
先在终端输入first查看需要输入的参数:
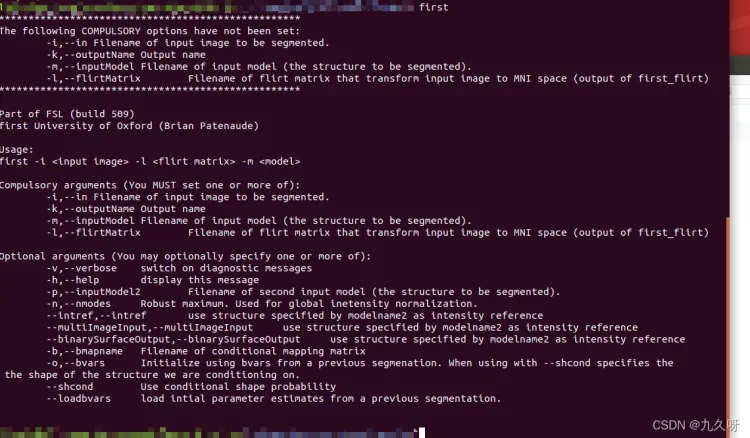
可以看到-i 要从哪个文件进行提取, -k输出分割文件名称,-m 分割的标准文件(*.bmv),-l
做flirt时的变换矩阵。
关于*.bmv文件:fsl自带的,在fsl安装目录data/first下面:
关于这两个文件夹有什么区别不清楚,但是海马在选中的文件夹中。
bash直接运行
flirt的变换矩阵即subjmat.mat,该文件在做配准的时候是需要用参数进行输出的,但是直接使用命令是默认不输出
命令:first -i subj.nii -l subjmat.mat -m L_Hipp_bin.bmv -k l_hippo.nii
同样的方法生成右海马:
first -i subj.nii -l subjmat.mat -m R_Hipp_bin.bmv -k r_hippo.nii
使用freesurfer中的mri_concat命令将左海马和右海马合并成整个海马体:
mri_concat –combine l_hippo.nii r_hippo.nii –o hippo.nii
得到hippo.nii就是海马文件:
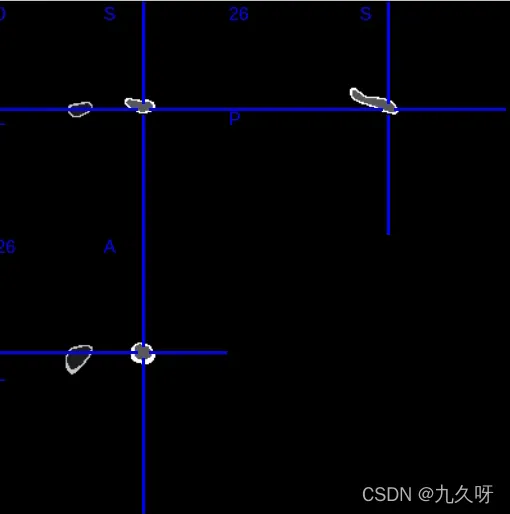
python+nipype实现
分割成左右海马体+合并代码
1 | import os |
1 | first -i ADNI_002_S_0619_affine.nii.gz -l subject.mat -m /usr/local/fsl/data/first/models_317_bin/L_Hipp_bin.bmv -k l_hippo.nii |