Author: haoransun
Wechat: SHR—97
目录
- 下载DICOM格式数据
- DICOM -> NIFTI格式 输出s开头文件
- 时间层校正Slice Timing 输出as开头文件
- 头动校正Realignment 输出ras开头文件
- 归一化Normalize 输出w开头文件
- 平滑Smooth 输出sw开头文件
- 配准Co-register reslice
- 提取ROI
1. 下载DICOM格式数据
登录地址:
https://ida.loni.usc.edu/ladvq_search.xjsp?project=ADNI&page=SEARCH&subPage=ADV_QUERY
输入登录账号及密码->ADNI GO->DOWNLOAD IMAGE Collections->Advanced Search(beta)->
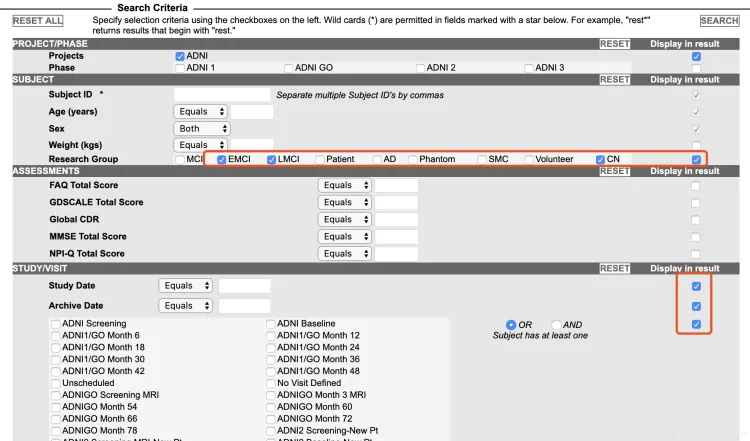
确定Research Group: EMCI LCMI AD CN
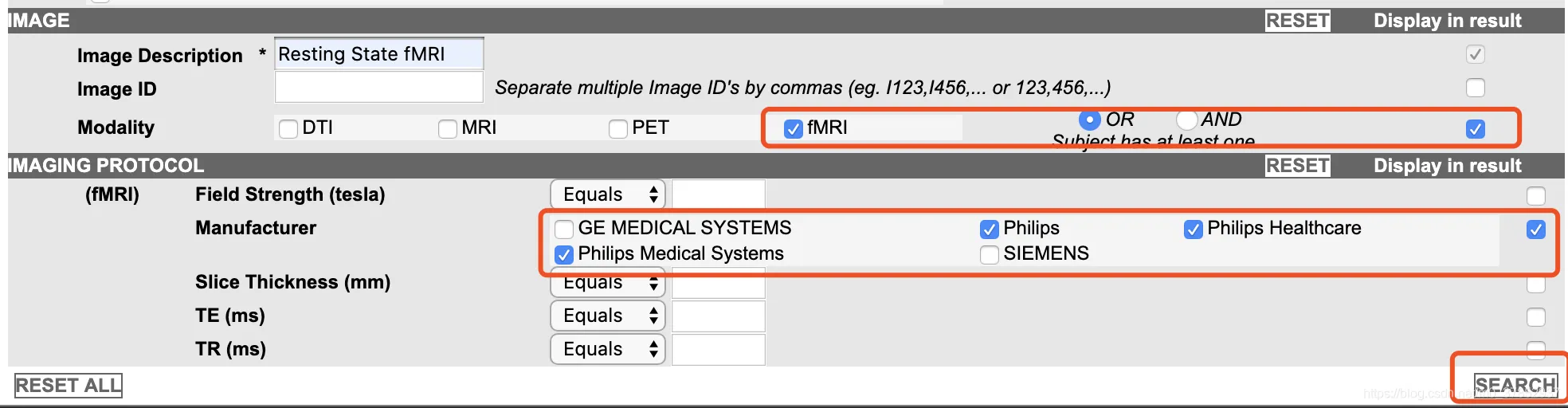
Image Description:Resting State fMRI-> Display in result->csv download (注:网页加载很慢)
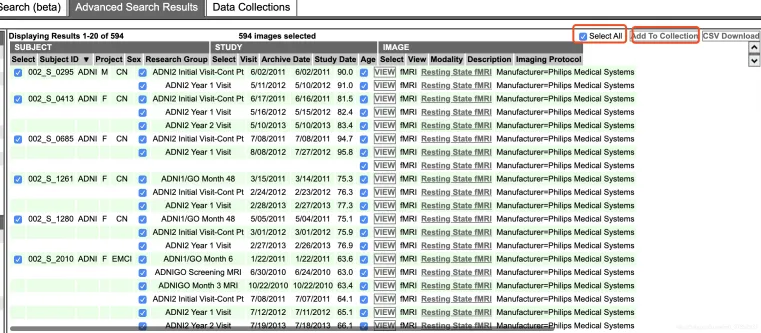
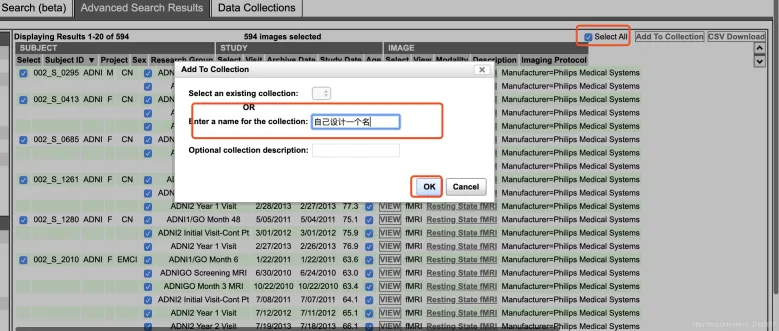

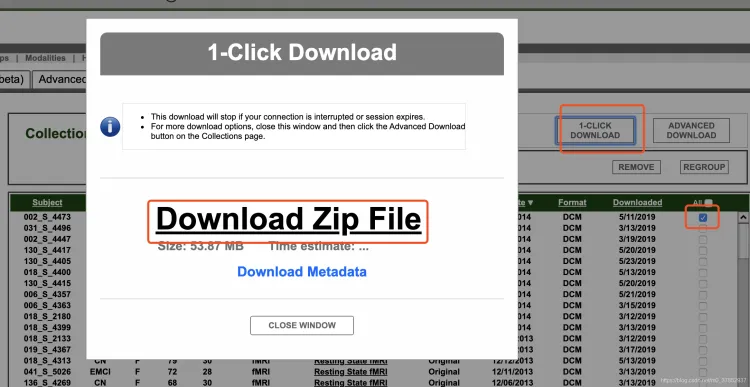
建议单个下,如果all同时下,会出现断网,网中断,下载失败或文件不全。且不好统计哪些是下载过的**。**
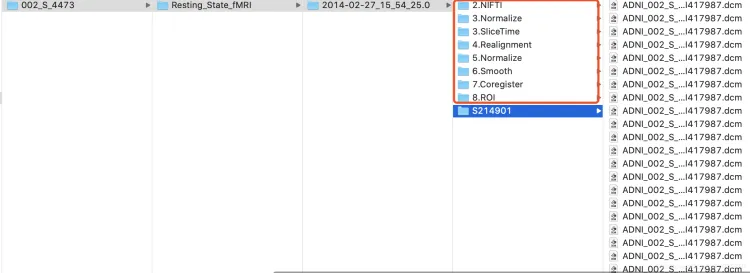
已知下载好的文件格式解压如下(注:文件必须是6720个dicom,如果少了,说明网络中断导致文件没下载全,需要重新下载。):
事先在每个subject里存好每个步骤对应路径(可以用程序在每个人对应路径下创建以下文件夹):
1 | import os |
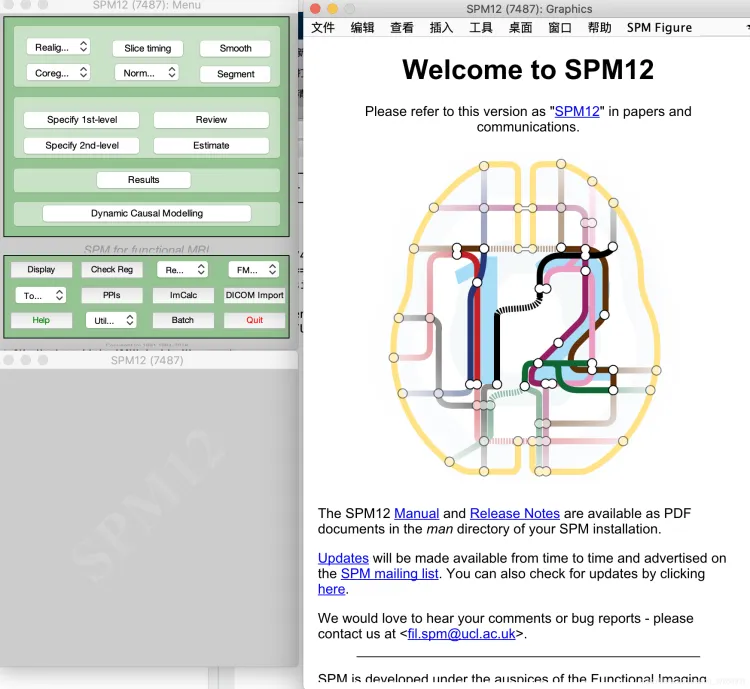
Matlab 配好spm12 环境,输入spm(以下三个窗口都不要随意叉掉,后面要用到)
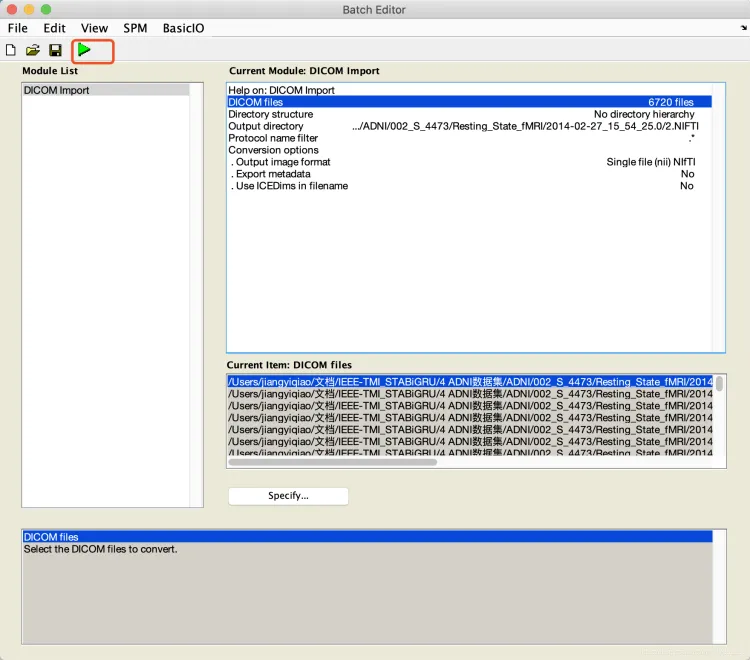
Dicom import

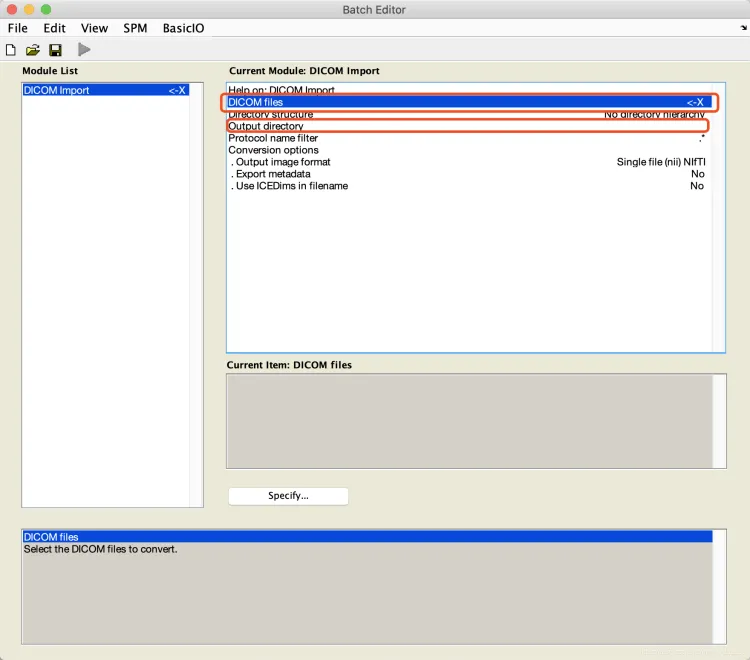
文件输入选中6720个文件
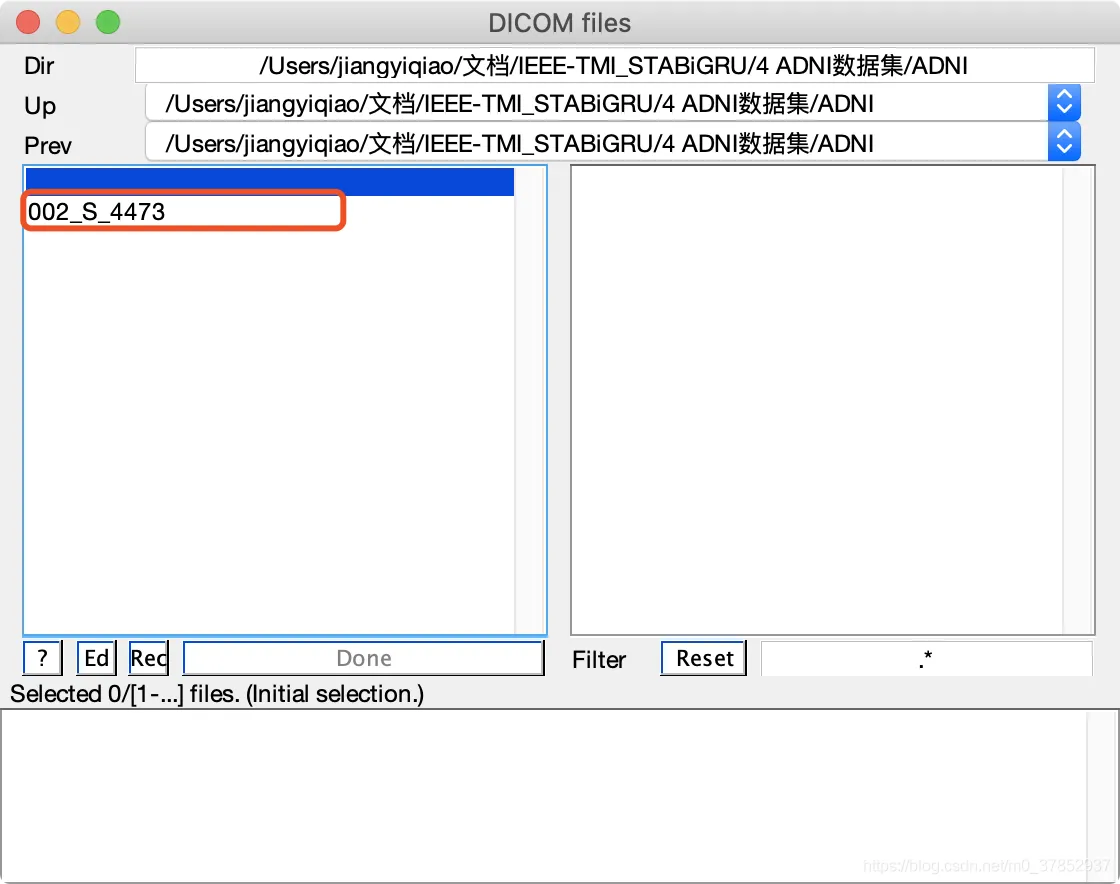
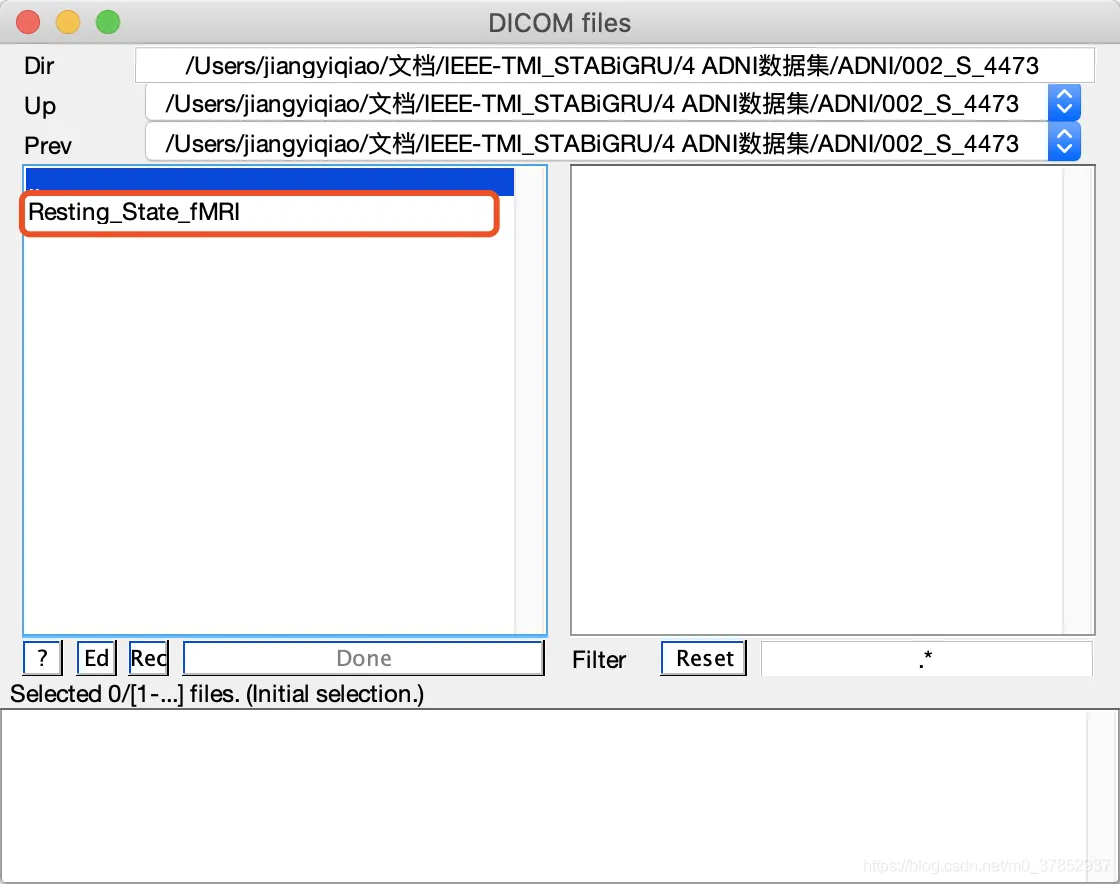
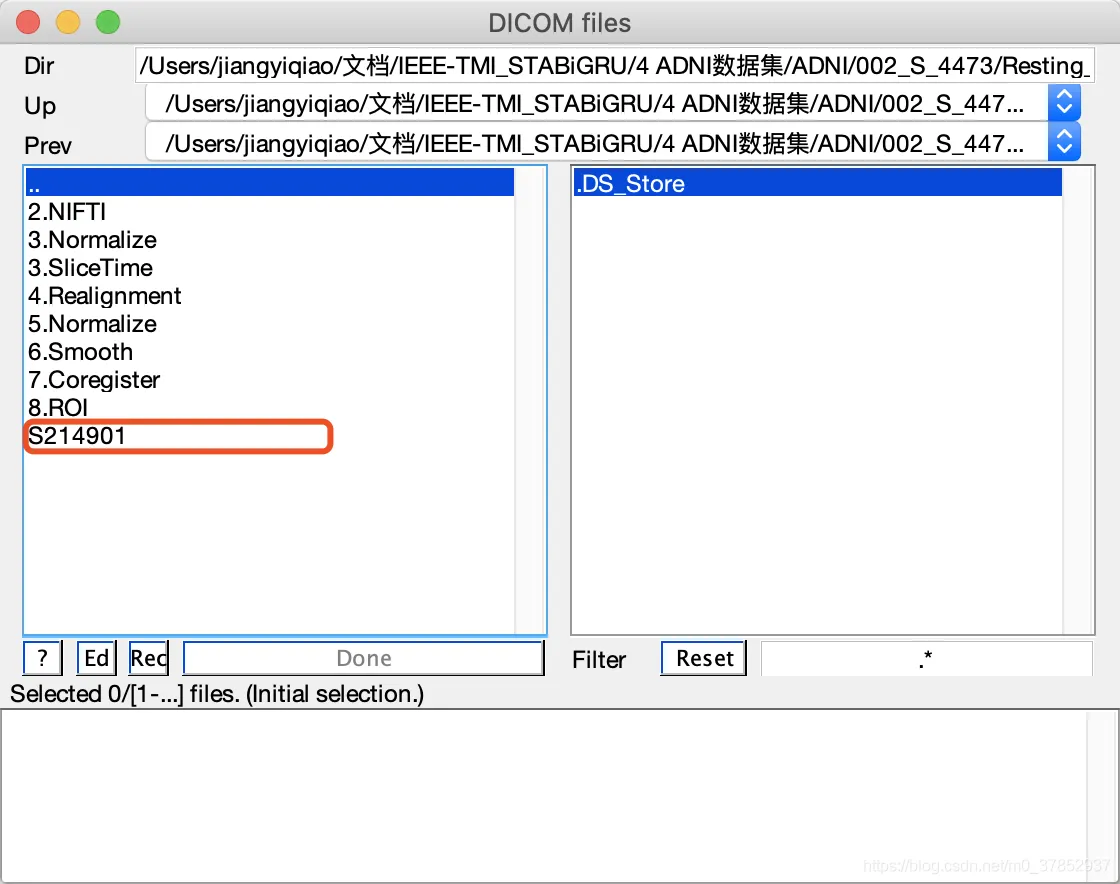
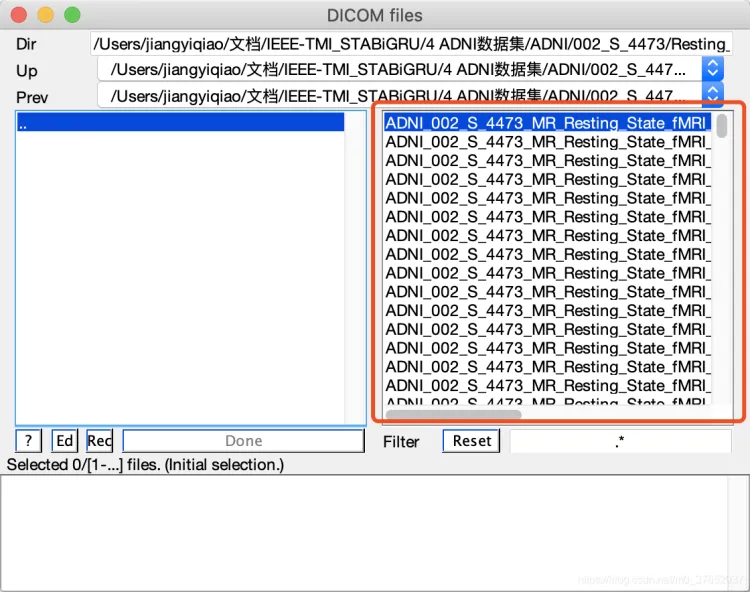

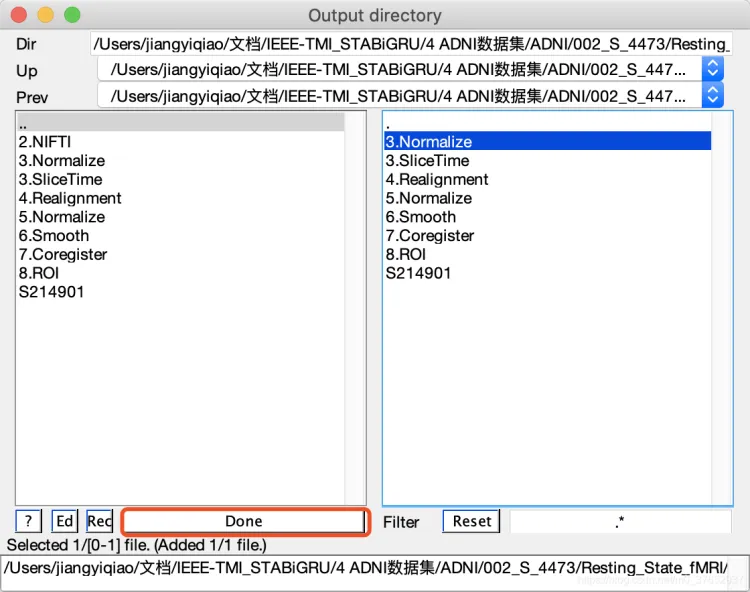
文件输出保存路径
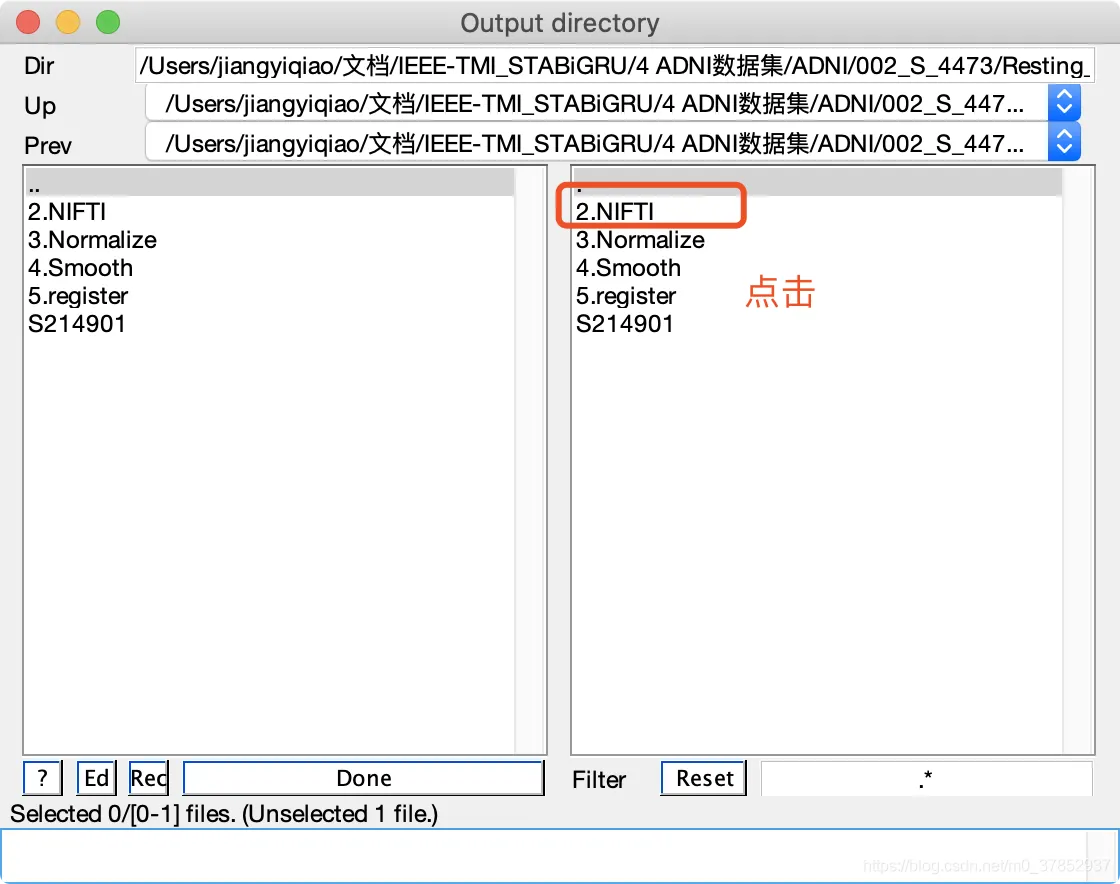
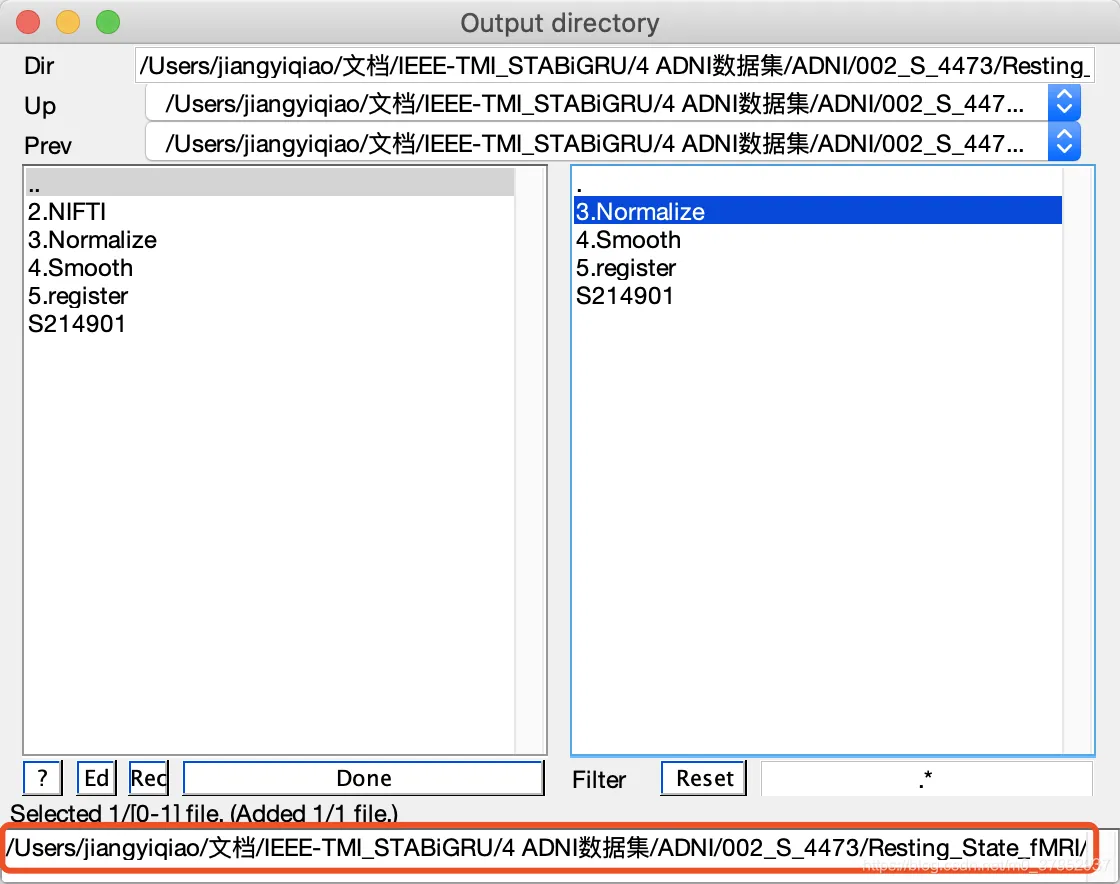
如果点错了,想要重选,点击红框以选中路径,文件路径就会消失,重选即可)
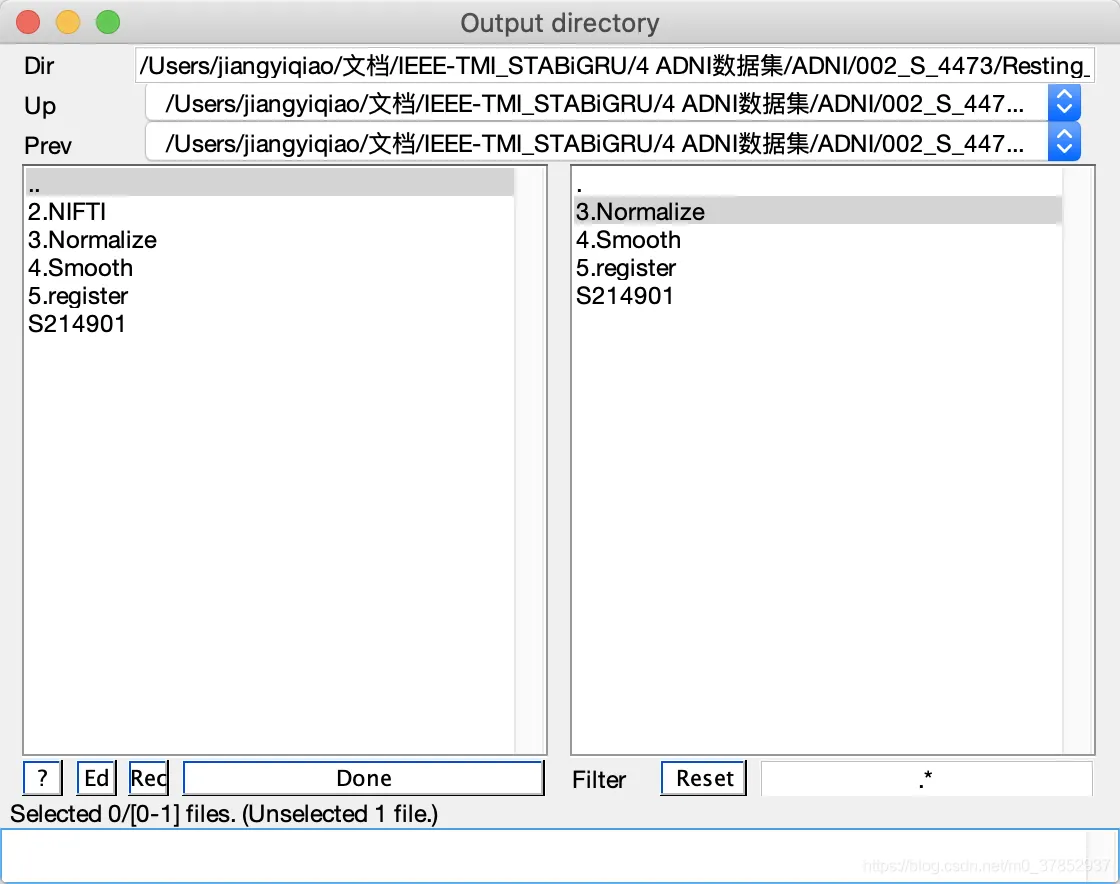
选好后点击done:
想要批量处理,可以先直接把第一个没加文件的Module List复制
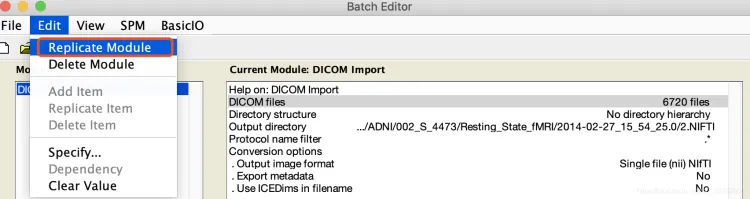
想要批量处理,可以先直接把第一个Module List 复制
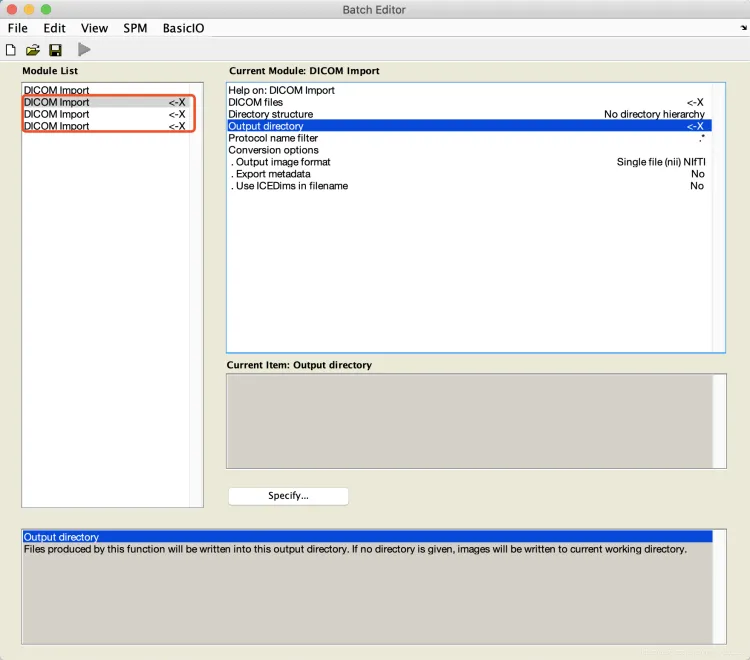
显示处理进度,耗时5分钟
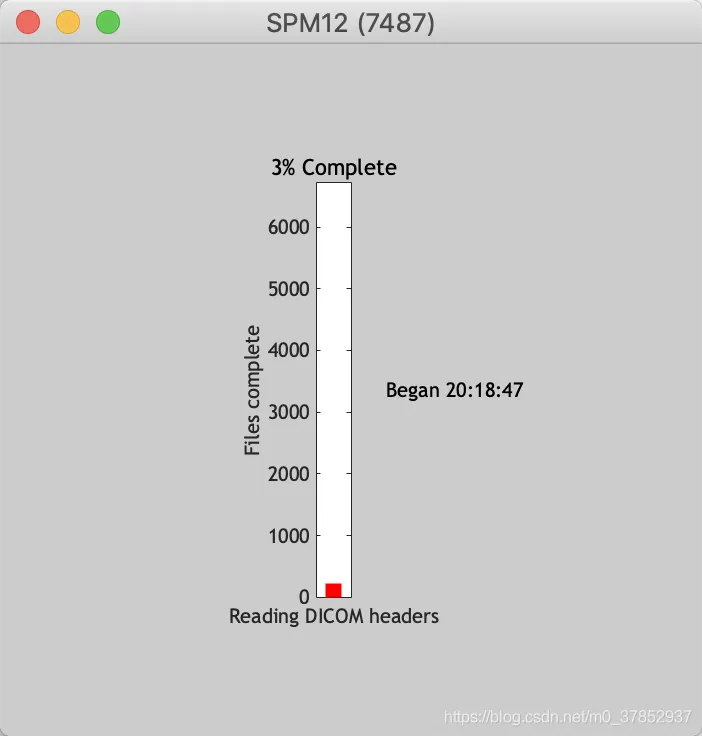
有时候源文件生出结果只有一个文件,这个subject是不可用的,
2. DICOM -> NIFTI格式 输出s开头文件
原始的多个dicm文件得到140的nii文件
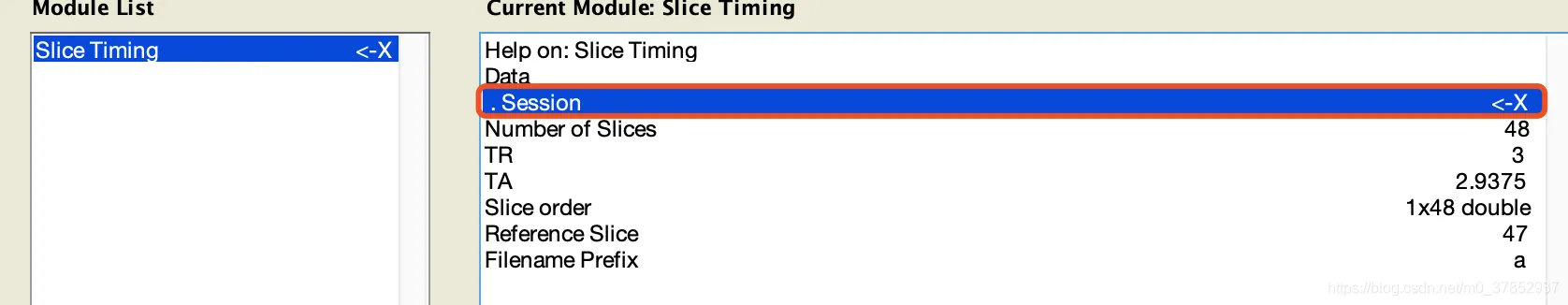
3. 时间层校正Slice Timing 输出as开头文件
Number pf slice: 48
TR: 3
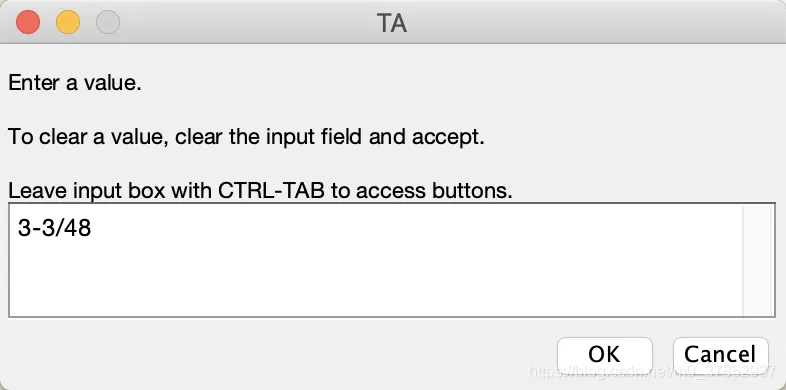
TA: 3-3/48
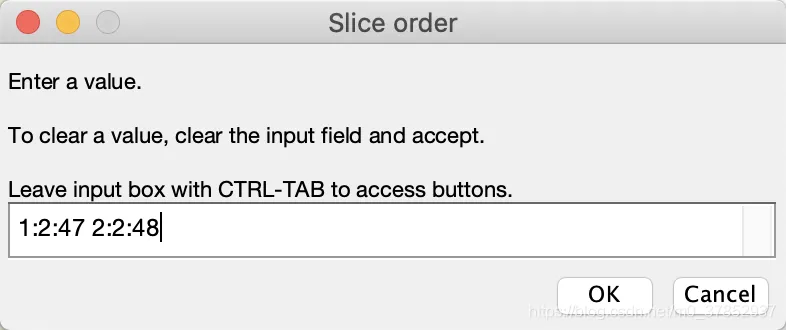
Slice order:输入修改参数 1:2:47 2:2:48
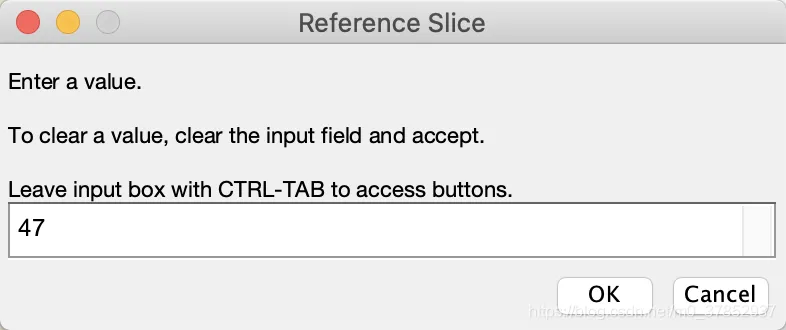
Reference Slice:输入47(这里也可以是24,一般取48/2=24 中间较好)
选择上一步骤的140个文件,设置参数后运行
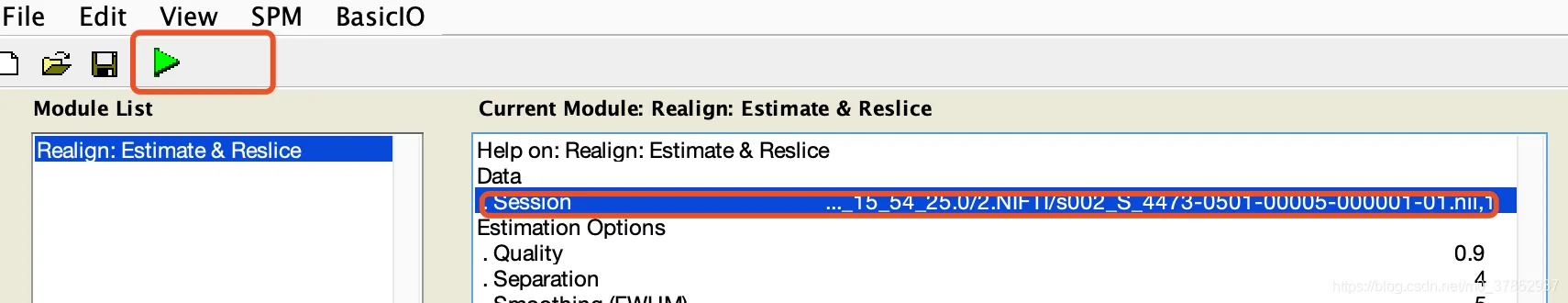
4. 头动校正Realignment 输出ras开头文件

输入上一步骤得到的140个文件,没有参数设置,直接运行
运行后会得到如下头动文件,自行记录和判定即可。
得到的中间图查看是否符合规范
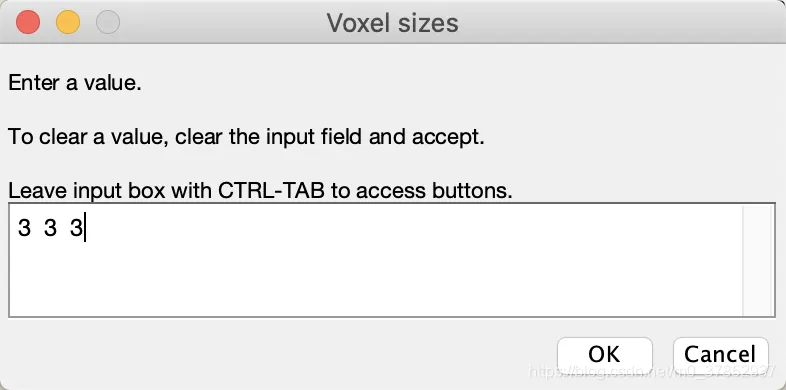
5. 归一化Normalize 输出w开头文件
修改Voxel sizes—[改为3 3 3]

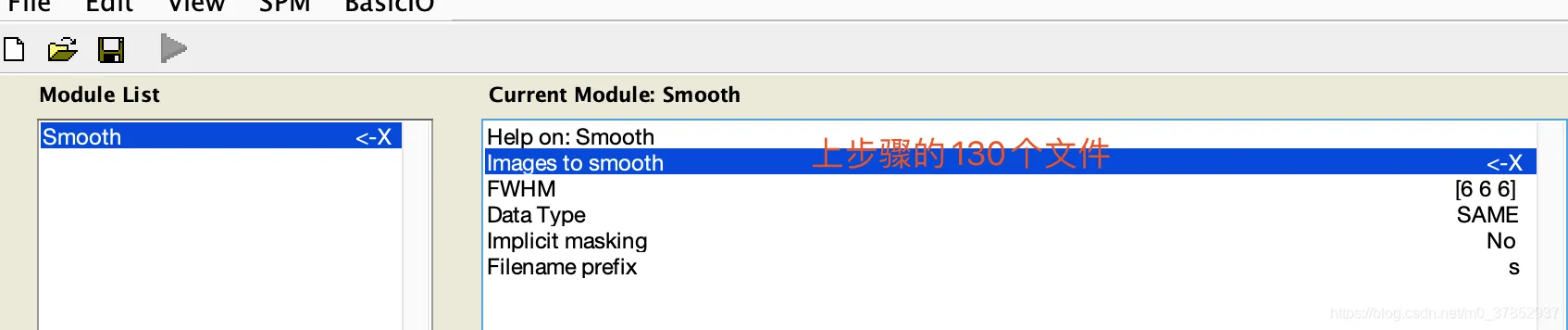
6. 平滑Smooth 输出sw开头文件
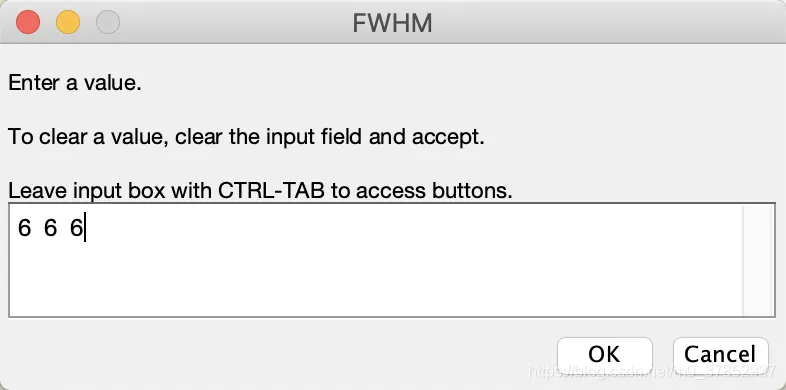
将默认的[8,8,8],改成[6,6,6]
7. 配准Co-register reslice
选择Coregister(Reslice)
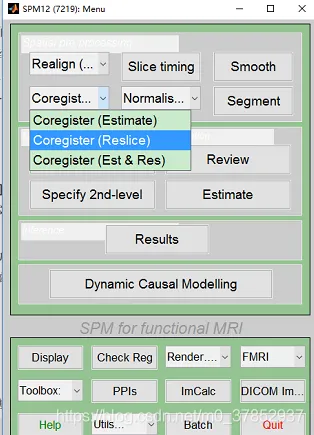


然后点击运行。运行完成后,将得到的rswaf开头的130个新文件。
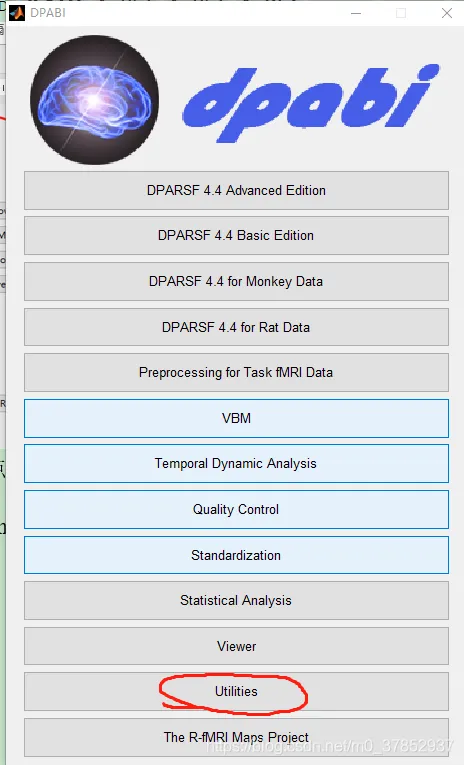
8. 提取ROI
在matlab里输入dpabi,运行dpabi。在出现的可视化操作界面中,按下图顺序点击按钮
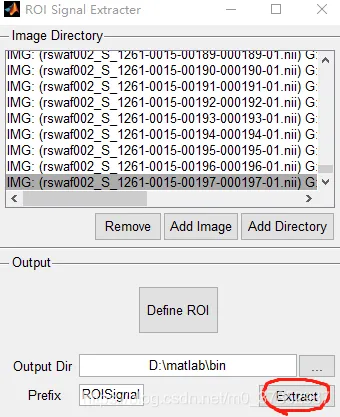
在出现的界面中,点击Add image 添加rswaf开头的187个文件
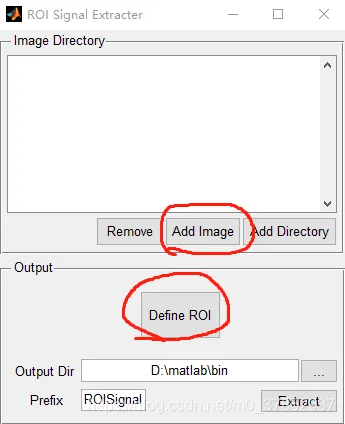
点击Define ROI 选择AAL atlas,然后点OK

然后点击Output Dir 来修改输出路径,接着点击Extract

运行完成后,在定义的输出路径下有130*7=910个文件
其中ROISignals开头的(130*2个文件)就是需要的fmri时间序列文件,
共包含txt和mat两种格式,两种格式文件所存内容一致,只是文件格式不一样。
可以使用BrainNetView等工具进行绘制(有待验证)
ROISignals开头的每个mat文件,储存了1×116的矩阵,表示该时间点下,116个脑区各自的数值。将130个mat文件按顺序组合成130*116的矩阵(可用matlab批处理完成),即是最终的数据矩阵。
130x116 去除了前10个时间序列节点,因为机器启动热加载导致的不平衡等问题**。**
AAL模板中 前90个为大脑脑区,研究中使用较多。后26个为小脑脑区,研究中使用较少。