Author: haoransun
Wechat: SHR—97

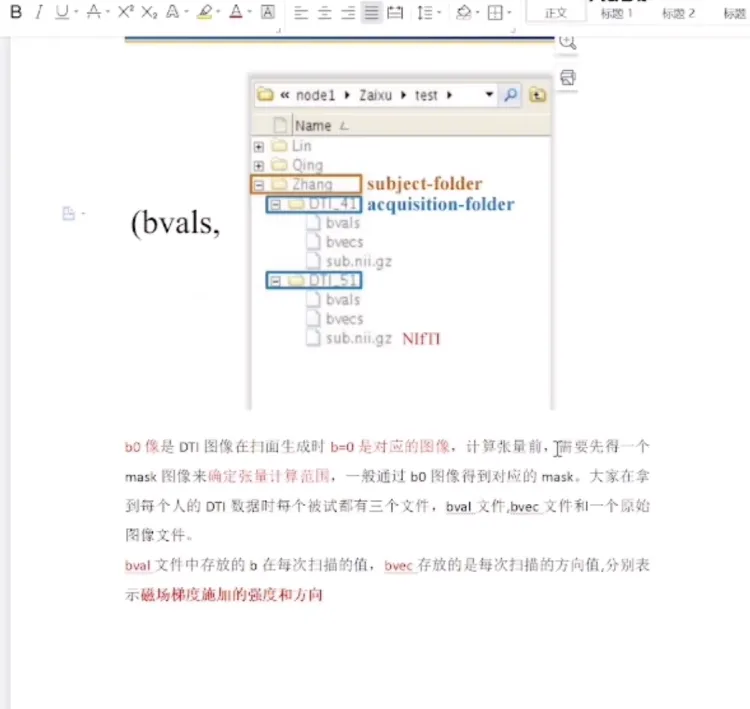
文件准备
设置一个CN/EMCI/LMCI文件夹,下面放入各个被试的数据

上面是pdf截图,具体细节查看pdf
第一步:文件夹的准备
dcm2nii.sh(无效)
https://www.jianshu.com/p/220f6f6f59ae
运行不成,使用软件做转换
1 | cd DTI_test |
第二步:提取b0
PANDA中使用extract_b0代码
1 | cd /home/pugongying/data/jupyterLab/new_dev/shr/Study5/CN/DTI_Result |
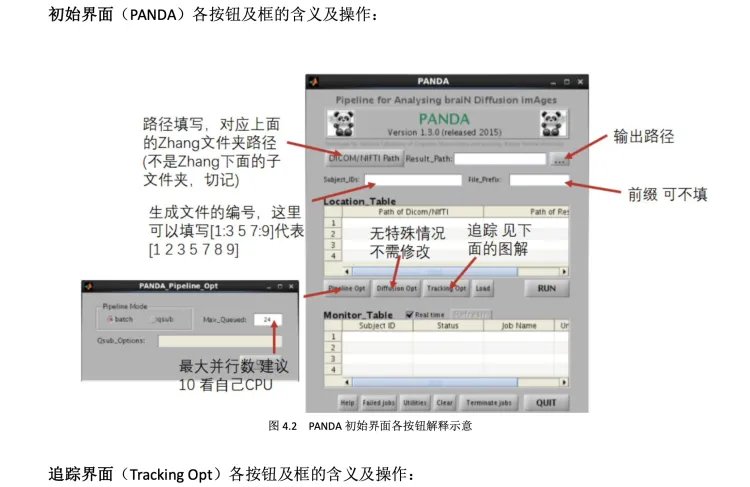
第三步:PANDA中点点点,进行预处理
选择DICOM–>NifTI
正式开始
第一步: 文件夹准备
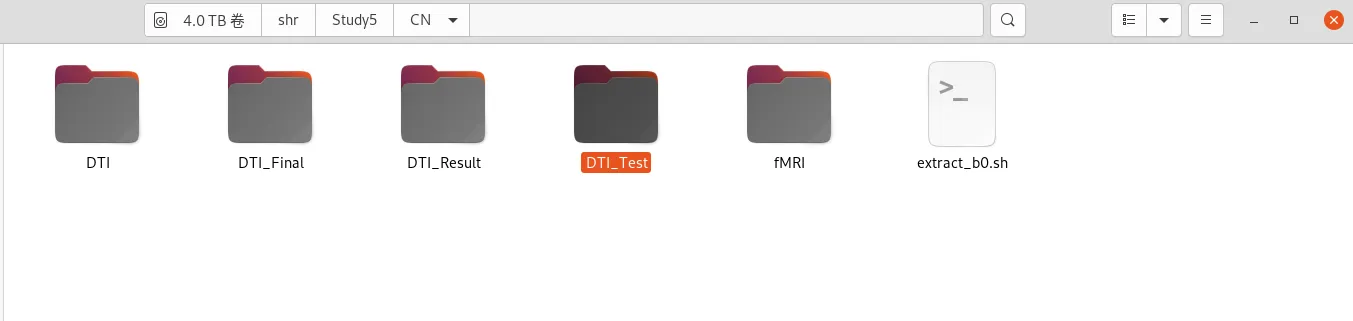
准备 CN/DTI_Test 文件夹
准备 CN/DTI_Result 文件夹
准备 CN/DTI_Final 文件夹
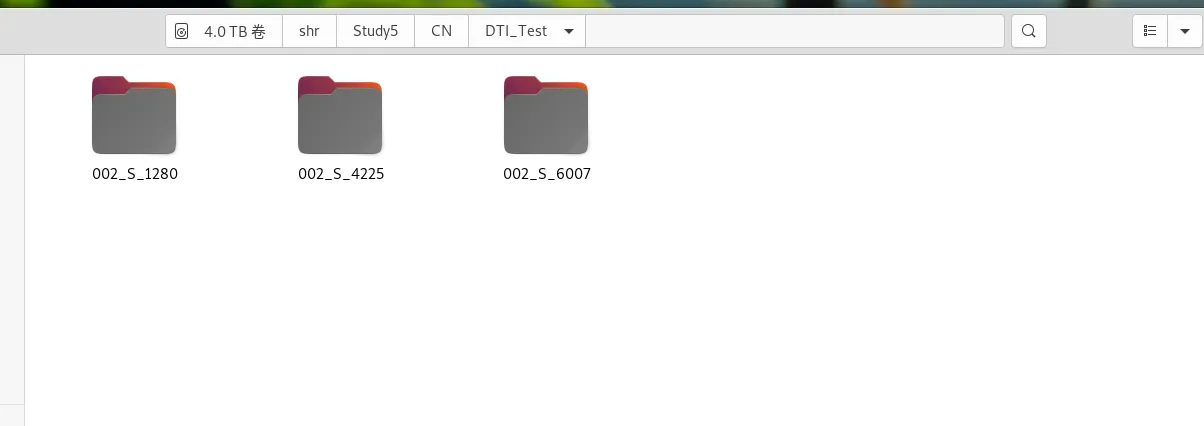
每个文件夹下放入对应受试者的文件夹名称,如下图所示
格式转换
选中DICOM -> NIFTI
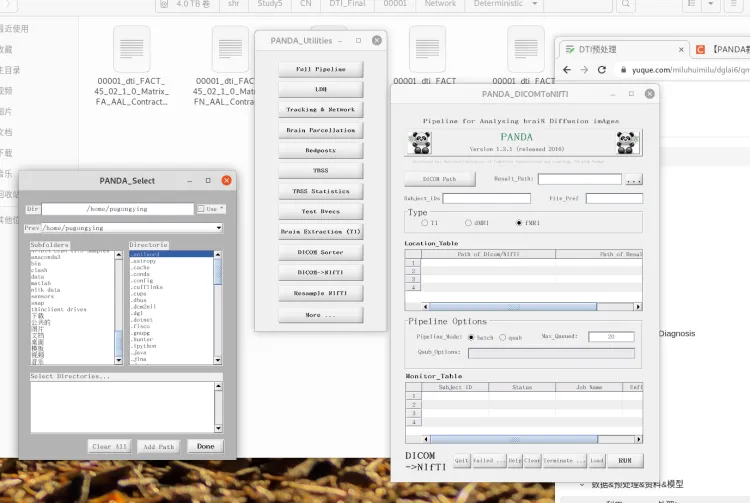
点击 DICOM Path 挑选Subject, 此处只选择了3个Subject
点击 Result Path 选择转换后图像保存路径,此处选择 CN/DTI_Result 文件夹
Subject_IDs 输入 1:3 因为选择了3个Subject
Type 选中 fMRI
点击RUN按钮, 进行格式转换
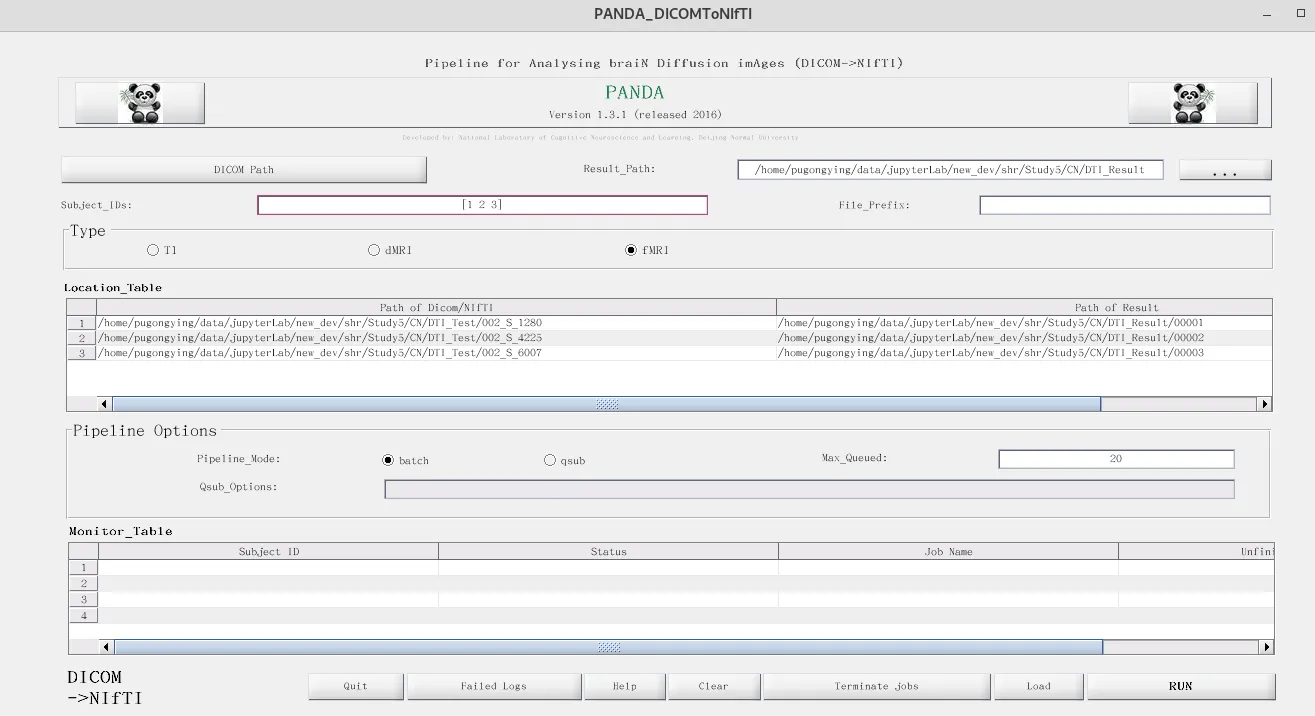
结果展示
CN/DTI_Result 文件夹 会出现每个Subject 对应的新编号,如 00001 00002 此处需要自己记录转换前后名称的变化
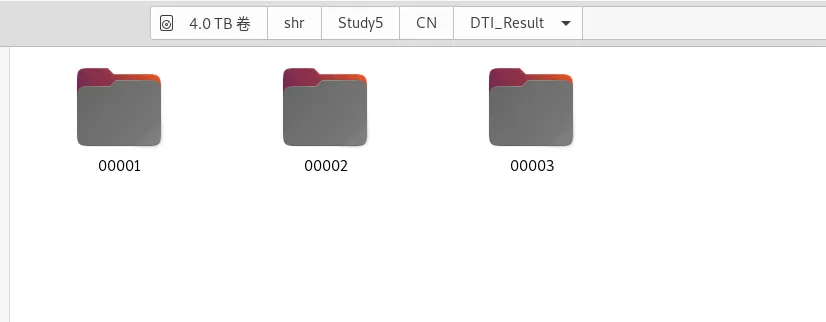
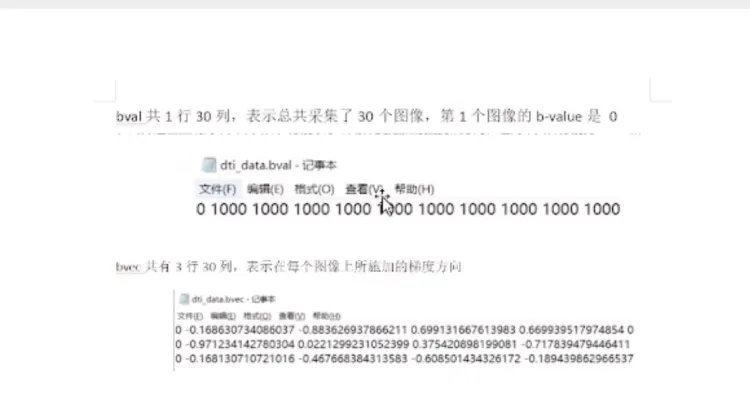
会出现3个文件,具体含义参见最上面
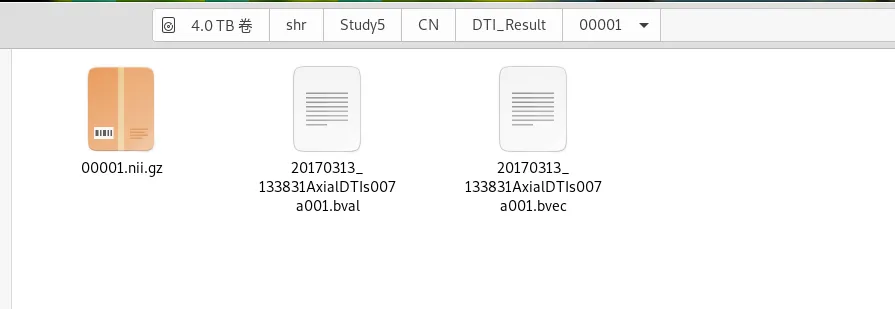
每一个Subject下新建一个名称为1的文件夹,将上面3个文件放入其中
其他Subject依次类比
第二步: 提取bo
1 | cd /home/pugongying/data/jupyterLab/new_dev/shr/Study5/CN/DTI_Result |
在CN文件夹路径下打开终端
运行该代码 sh extract_b0.sh
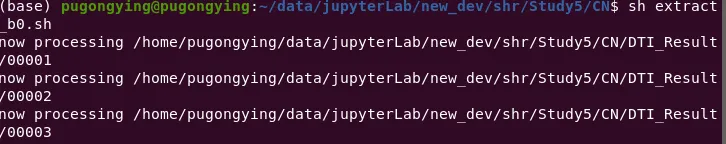
在 DTI_Result 文件夹下 出现 各个 Subject 对应的 b0文件
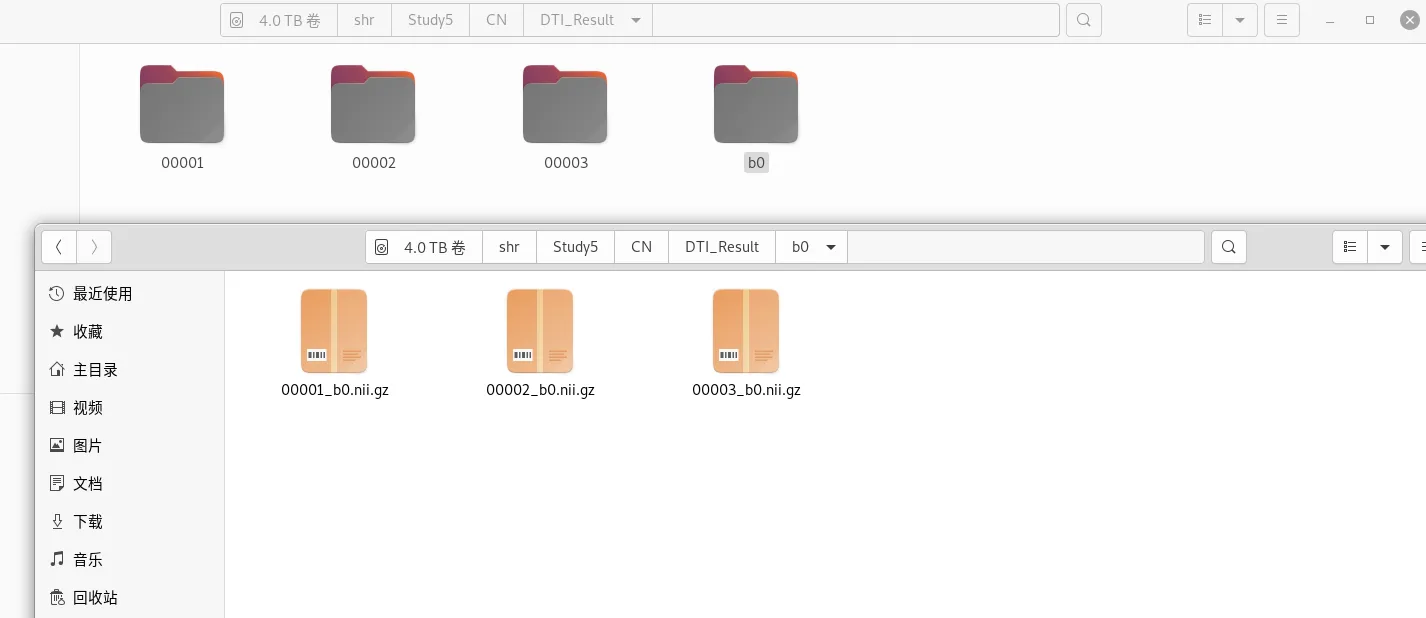
新建一个 b0文件夹, 将这3个b0文件放入其中,后面配准用
第三步: PANDA中点点点
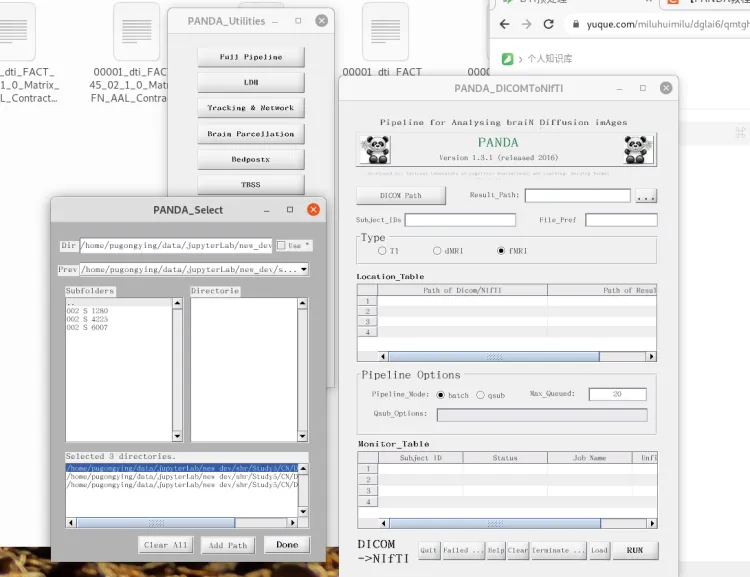
PANDA 中选择 DICOM/NIFTI Path, 挑选 DTI_Result 文件夹下的各个Subject
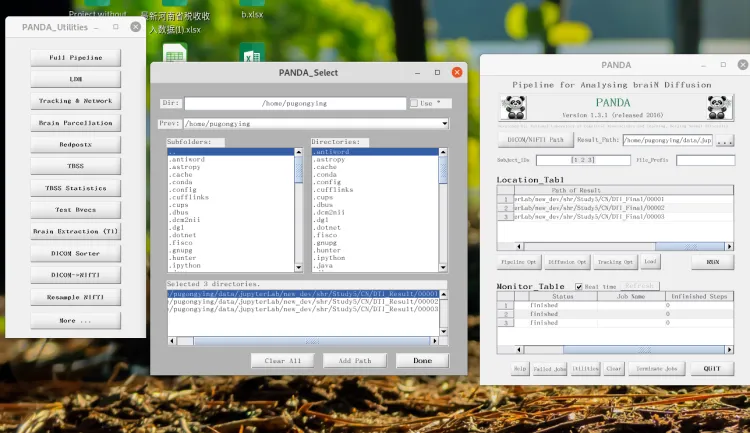
Result Path 选择 DTI_Final
Subject_IDs 输入 1:3 因为选择了3个Subject
Pipeline Opt 根据自己电脑情况进行设置
DIffusion Opt
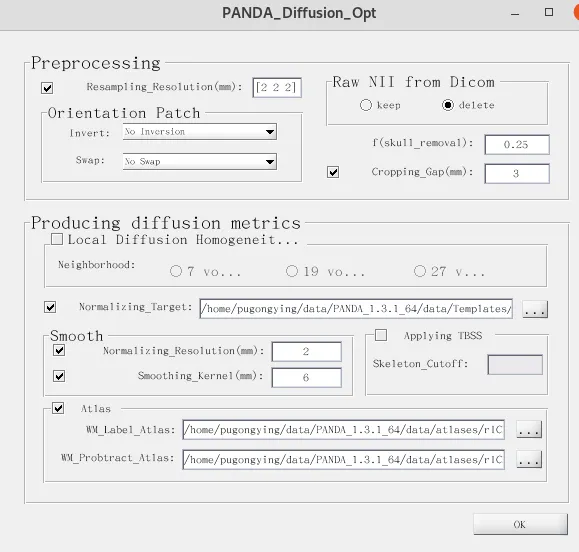
重采样到 2 2 2
头骨剥离 0.25
剪切图像 3
剩下默认按照图像所示,软件里面应该是默认填充这些选项
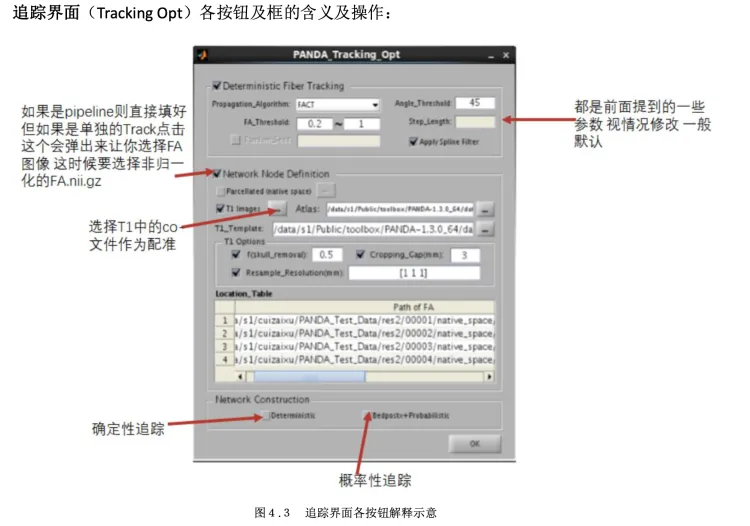
Tracking Opt
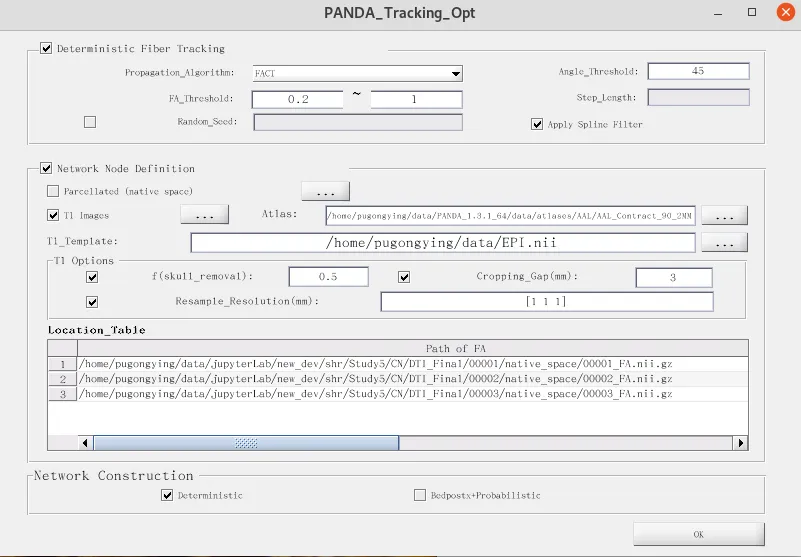
点击 T1 Image 后面的 … 按顺序选择 b0 里面的 bo.nii文件, 顺序要与刚开始输入Subject一致
Atlas 选择 PANDA 默认提供的 AAL 90 (默认填充)
T1_Template 选择 EPI.nii 模板文件
其他全部默认填充
Network Construction 选择 左边确定性网络, 右边的概率网络耗费时间漫长
结果展示
DTI_Final文件夹如下
点击 Network - Deterministic
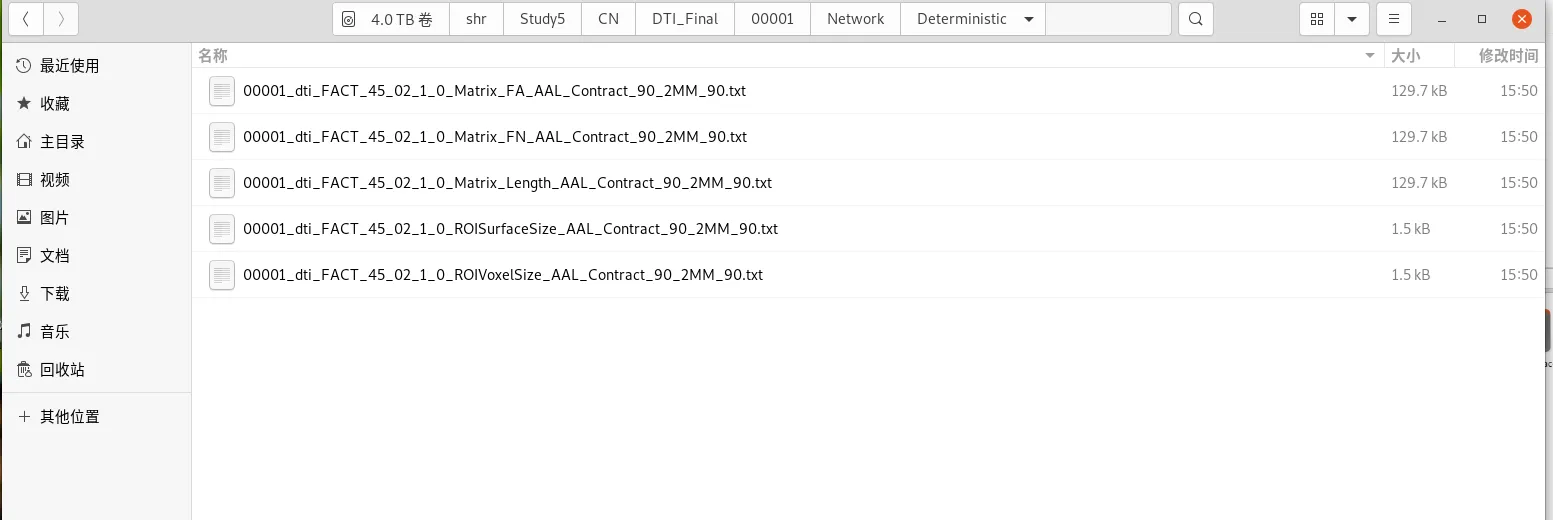

我们选择 FA 或者 FN 值